王一帆 陈子茵 贾新蕾 黄增朝 吕静 黄郁葱
摘 要:根据GenBank上登录的红笛鲷SR-BI基因设计引物,采用RT-PCR方法扩增该基因胞外段序列,然后将该基因胞外段序列定向克隆到原核表达载体pET-28a(+)中,将重组质粒转入大肠杆菌BL21进行IPTG诱导表达,并对重组表达菌株的表达条件进行优化。结果显示,成功构建了原核表达载体pET28a-SR-BI,并能在大肠杆菌BL21中表达,表达的融合蛋白分子量为50.0ku。融合蛋白的最佳诱导表达温度、IPTG浓度和时间分别为16℃、0.05mmol/L和8h。研究结果为进一步研究SR-BI的功能奠定了基础。
关键词:红笛鲷;SR-BI基因;原核表达;条件优化
中图分类号 S942.1文献标识码 A文章编号 1007-7731(2021)03-0001-06
Prokaryotic Expression and Condition Optimization of SR-BI Gene in Lutjanus sanguineus
WANG Yifan et al
(Fisheries College of Guangdong Ocean University/Provincial Key Laboratory of Pathogenic Biology and Epidemiology for Aquatic Economic Animals of Guangdong & Key Laboratory of Control for Diseases of Aquatic Economic Animals of Guangdong Higher Education Institute, Zhanjiang 524088, China)
Abstract:A pair of primers were designed based on Scavenger receptor class B type I(SR-BI) gene sequence published in GenBank. The extracellular region sequence of SR-BI gene was amplified by RT-PCR and then inserted into the pET-28a(+) vector to construct prokaryotic expression plasmid. The recombinant plasmid was transformed into E.coli BL21 to overexpressed in the presence of isopropyl-β-D-thiogalato pyranos ide (IPTG), and the expression conditions of the recombinant strain were optimized. The results showed that the prokaryotic expression plasmid pET28a-SR-BI was successfully constructed and expressed in E. coli BL21. The molecular weight of the expressed recombinant fusion protein was 50.0 ku. The optimal induction expression temperature, IPTG concentration and induction time of the recombinant fusion protein was 16℃, 0.05mmol/L and 8h, respectively. The research results laid the foundation for the further research on the functions of SR-BI.
Key words: Lutjanus sanguineus; SR-BI gene; Prokaryotic Expression; Condition optimization
清道夫受體(Scavenger Recptors,SRs)是一类与膜相关或可溶性的、能结合修饰后的LDL(Low-Density Lipoprotein)或其他多聚阴离子配体的跨膜糖蛋白[1],首次发现于小鼠动脉粥样硬化斑块的巨噬细胞上存在摄取和降解乙酰化低密度脂蛋白(ac-LDL)的结合位点,后来被称为巨噬细胞清道夫受体1[2]。根据分子结构、表达特征及与配体识别机制,目前已发现的清道夫受体至少包含8种不同类别(A、B、C、D、E、F、G、H)[1]。清道夫受体具有广泛的受体识别谱,能够识别修饰低密度脂蛋白、革兰氏阴性菌细胞壁成分的脂多糖(LPS)和革兰氏阳性菌细胞壁成分磷壁酸(LTA)多种微生物结构成分,还可识别由细胞膜内转运到膜外的磷脂酰丝氨酸,并能够结合多种蛋白质、核苷酸、多糖以及脂类等,参与机体一系列的生理和病理过程,包括动脉粥样硬化形成或抑制、阿尔茨海默氏病、宿主免疫防御、细胞粘附、凋亡细胞清除、组织保护和维护机体平衡等[3-5],在动物生命代谢过程中起着重要作用。
清道夫受体BI(Scavenger receptor class B type I,SR-BI)是哺乳动物中研究较为深入的清道夫受体基因家族成员之一,与CD36和LIMPⅡ(lysosomal integral membrane protein Ⅱ)同属B族清道夫受体[3]。SR-BI为典型的Ⅲ型跨膜糖蛋白,结构如马蹄样,由5个区域构成:1个大的环状细胞外域、2个胞内域、2个跨膜域,胞外域中间部分是主要的配体结合区域。SR-BI是目前在分子水平上证实的第一个高密度脂蛋白(high density lipoprotein,HDL)受体,介导脂质的选择性摄取和胆固醇的逆转运[6]。近年来,越来越多的研究表明,SR-BI不仅参与脂类代谢和与某些代谢性疾病的形成有关外,并且作为模式识别受体在机体先天性免疫中也发挥着重要作用[7-8]。SR-BI作为一种具有重要功能的模式识别受体,能够识别许多病原相关分子模式,如LPS[9]、革兰氏阴性菌(大肠杆菌、沙门氏菌)和革兰氏阳性菌(金黄色葡萄球菌、李斯特氏菌)[10]及结核杆菌[11]等,因此作为吞噬性受体直接介导非调理素病原微生物的吞噬或者与TLR(Toll-like receptors)形成共受体调节促炎症反应因子共同应答病原微生物,在对外来病原,特别是对细菌及其炎症反应复合物的免疫应答中发挥关键作用。此外,有研究表明,SR-BI作为(hepatitis C virus,HCV)进入宿主肝细胞的一个可能的靶受体,与丙型肝炎病毒的入侵和感染有关[12-13]。
目前,在水生动物的相关研究较少,目前仅有大西洋鲑(Salmo salar L.)[14]、日本沼虾(Macrobrachium nipponense)[15]、日本囊對虾(Marsupenaeus japonicus)[16]和菲律宾帘蛤[17]等水生动物的SR-BI基因得到克隆鉴定,研究发现水生动物SR-BI与哺乳动物SR-BI具有相似的结构特征,参与脂质代谢和抗菌免疫,但对于鱼类的SR-BI的功能尚未有相关的研究报道。红笛鲷(Lutjanus sanguineus)是我国南方沿海地区重要的海水养殖鱼种,然而随着集约化养殖规模的不断扩大和养殖环境日益恶化,病害频繁暴发,造成了巨大的经济损失[18]。目前,对于红笛鲷的免疫系统研究尚处起步阶段,已获得了红笛鲷的SR-BI基因全长,但尚未见相关的蛋白水平研究。系统研究红笛鲷SR-BI基因的功能,对于阐明鱼类的脂类代谢、免疫识别等重要生理机制具有重要意义。为此,本研究通过构建SR-BI蛋白胞外段基因的原核表达载体,将其转入到原核表达载体BL21中进行SR-BI重组蛋白的表达,对诱导温度、IPTG浓度和诱导时间等表达条件进行优化,并对SR-BI蛋白进行纯化和鉴定,以期为进一步研究红笛鲷SR-BI的功能奠定基础。
1 材料与方法
1.1 材料 试验用健康红笛鲷(体质量约500g)购自湛江某海水养殖场;原核表达载体pET-28a(+)、大肠杆菌BL21由本实验室保存;总RNA提取试剂盒(TriZolTM Up Plus RNA Kit)、cDNA反转录试剂盒(TransScript? One-Step gDNA Removal and cDNA Synthesis SuperMix)购自北京全式金生物技术公司;质粒提取试剂盒(GeneJET Plasmid Miniprep Kit)、琼脂糖凝胶回收试剂盒(GeneJET Gel Extraction Kit)购自赛默飞世尔科技(Thermo Fisher Scientific)公司;引物、氨苄西林(Amp+)、异丙基硫代-β-D-半乳糖苷(IPTG)购自上海生工生物工程有限公司;考马斯亮蓝快速染色液、一抗、二抗购自天根生化公司。其他试剂均为分析纯。
1.2 方法
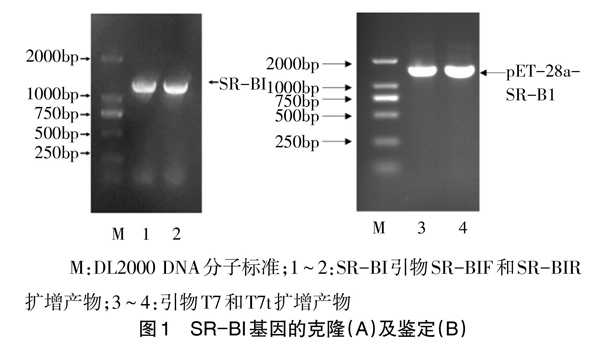
1.2.1 SR-BI基因的扩增 在无菌条件下取红笛鲷的脾、头肾组织,加入液氮研磨组织,于无RNA酶操作环境下参总RNA抽提试剂盒说明书提取组织总RNA。1.2%琼脂糖凝胶电泳检测总RNA的完整性,紫外分光光度法检测其纯度和浓度,确保其OD260/OD280在1.8~2.0。组织的总RNA按照cDNA反转录试剂盒说明书合成cDNA第一链,置-40℃保存待用。根据笔者提交NCBI的红笛鲷SR-BI基因胞外段序列(GenBank登录号:MW246167)设计含EcoRI和XhoI酶切位点上下游引物F/R,上游引物F:CCGGAATTCATGGACGACCAGATCGTTAAAAAC,下游引物R:CCGCTCGAGTTATTCCATAACAGCCGGC。PCR引物由生工生物工程(上海)股份有限公司合成。以cDNA作为模板扩增SR-BI蛋白的胞外段基因序列,PCR扩增程序为:94℃、3min,94℃、30s,60℃、30s,72℃、60s;35个循环,循环结束后72℃延伸6min。PCR反应结束后其产物用1.2%琼脂糖凝胶电泳检测,扩增的目的片段条带按切胶回收试剂盒说明书进行切胶回收,回收产物与pMD-18T 载体连接并转化DH5α感受态,涂布于氨苄青霉素(AMP)浓度为100μg/mL的LB琼脂培养基上,挑取单克隆并进行菌落PCR鉴定,阳性克隆送至生工生物工程(上海)股份有限公司进行测序。
1.2.2 原核表达载体pET28a-SR-BI的构建 将序列测序正确的pMD-18T-SR-BI重组质粒与原核表达空质粒pET28a参质粒提取说明书分别提取质粒,然后用限制性内切酶EcoRI和XhoI分别进行双酶切,37℃温水浴中2~3h后,将酶切产物分别进行琼脂凝胶电泳和切胶回收。将纯化后的酶切产物利用T4 DNA连接酶连接到表达质粒pET28a构建成重组质粒pET28a-SR-BI,然后转入大肠杆菌BL21,涂布于Kana+的LB琼脂培养基上,进行菌落PCR鉴定,阳性克隆送至生工生物工程(上海)股份有限公司进行测序,确保构建的表达序列不突变和移码。
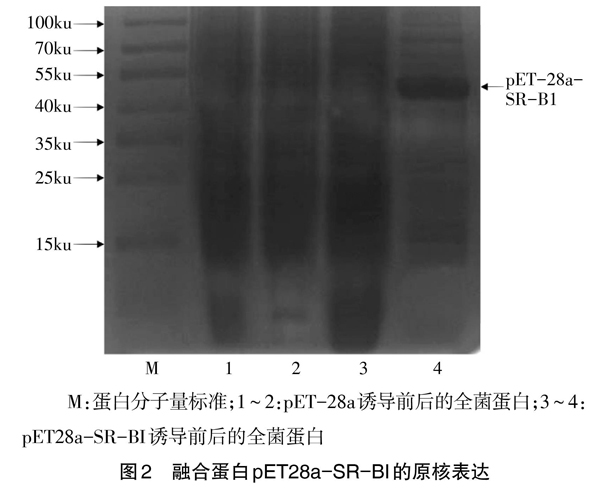
1.2.3 重组蛋白诱导表达 将含有重组质粒pET28a-SR-BI和空质粒pET28a的BL21分别接种于含100μg/mLKana+的LB中,37℃、200r/min振荡培养过夜,将活化的菌种按照1:100的比例接种于含100μg/mLKana+的LB中,37℃、200r/min振荡培养至吸光度(OD600)达0.4~0.6,然后加入0.5mmol/L的诱导剂异丙基硫代半乳糖苷(IPTG)进行诱导表达6h,取1mL菌液12000g离心1min收集菌液,使用无菌PBS洗涤3次,加入50μL的PBS悬浮菌体,按照1∶5比例加入6×Loading Buffer混匀,100℃煮沸5min,离心后取上清进行十二烷基硫酸钠聚丙烯酰胺凝胶电泳(SDS-PAGE)分析。
1.2.4 原核表达的条件优化
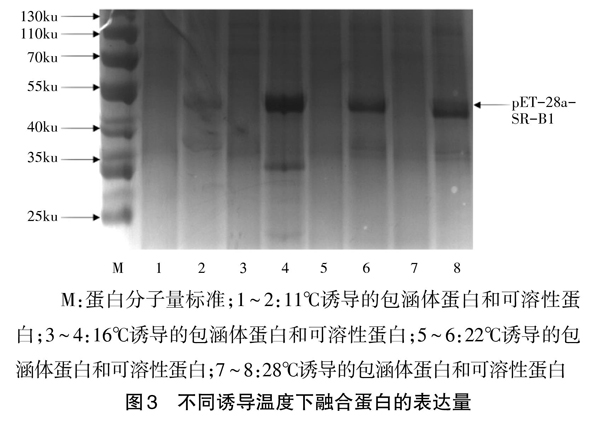
1.2.4.1 温度 IPTG诱导浓度设置为0.1mmol/L,分别于11℃、16℃、22℃和28℃恒温条件下200r振荡培养6h,分别离心收集菌体,用PBS清洗菌体后进行超声破碎。超声波细胞粉碎机的程序设置为:功率为200w,破碎行6s,间隔8s,破碎至菌液澄清透亮,高速离心分离上清和沉淀,SDS-PAGE分析目的蛋白的表达量。
1.2.4.2 IPTG浓度 IPTG诱导浓度分别设置为0.05、0.1、0.4和0.7mmol/L,16℃、200r/min振荡培养6h后分别取样,分别离心收集菌液,蛋白提取和SDS-PAG方法同1.2.4.1。
本研究构建了的原核表达载体pET28a-SR-BI,并在大肠杆菌中成功表达,获得了融合蛋白最佳诱导表达温度、IPTG诱导浓度和诱导时间分别为16℃、0.05mmol/L和8h,表达的融合蛋白分子量为50.0ku。
参考文献
[1]Plüddemann A,Neyen C,Gordon S.Macrophage scavenger receptors and host-derived ligands[J].Methods,2007,43(3):207-217.
[2]Goldstein J L,Ho Y K,Basu S K,et al.Binding site on macrophages that mediates uptake and degradation of acetylated low density lipoprotein,producing massive cholesterol deposition[J].Proceedings of the National Academy of Sciences of the United States of America,1979,76(1):333-337.
[3]Murphy J E,Tedbury P R,Homer-Vanniasinkam S,et al.Biochemistry and cell biology of mammalian scavenger receptors[J].Atherosclerosis,2005,182(1):1-15.
[4]Areschoug T,Gordon S.Scavenger receptors:role in innate immunity and microbial pathogenesis[J].Cellular microbiology,2009,11(8):1160-1169.
[5]Canton J,Neculai D,Grinstein S.Scavenger receptors in homeostasis and immunity[J].Nature reviews.Immunology,2013,13(9):621-634.
[6]Acton S,Rigotti A,Landschulz K T,et al.Identification of scavenger receptor SR-BI as a high density lipoprotein receptor[J]. Science,1996,271(5248):518-520.
[7]董占鵬,柴春利,代方银,等.家蚕B型清道夫受体基因的鉴定及系统发生与表达分析[J].蚕业科学,2010,36(05):743-753.
[8]Peiser L,Mukhopadhyay S,Gordon S.Scavenger receptors in innate immunity[J].Current opinion in immunology,2002,14(1):123-128.
[9]Vishnyakova T G,Bocharov A V,Baranova I N,et al.Binding and internalization of lipopolysaccharide by Cla-1,a human orthologue of rodent scavenger receptor B1[J].The Journal of biological chemistry,2003,278(25):22771-22780.
[10]Vishnyakova T G,Kurlander R,Bocharov A V,et al.CLA-1 and its splicing variant CLA-2 mediate bacterial adhesion and cytosolic bacterial invasion in mammalian cells[J].Proceedings of the National Academy of Sciences of the United States of America,2006,103(45):16888-16893.
[11]Philips J A,Rubin E J,Perrimon N.Drosophila RNAi screen reveals CD36 family member required for mycobacterial infection[J].Science,2005,309(5738):1251-1253.
[12]Dubuisson J,Helle F,Cocquerel L.Early steps of the hepatitis C virus life cycle[J].Cellular microbiology,2008,10(4):821-827.
[13]Lavillette D,Tarr A W,Voisset C,et al.Characterization of host-range and cell entry properties of the major genotypes and subtypes of hepatitis C virus[J].Hepatology,2005,41(2):265-274.
[14]Kleveland E J,Syvertsen B L,Ruyter B,et al.Characterization of scavenger receptor class B,type I in Atlantic salmon (Salmo salar L.) [J].Lipids,2006,41(11):1017-1027.
[15]Ding Z,Luo N,Kong Y,et al.Scavenger Receptor Class B,Type I,a CD36 Related Protein in Macrobrachium nipponense:Characterization,RNA Interference,and Expression Analysis with Different Dietary Lipid Sources[J].International journal of genomics,2016,6325927.
[16]Bi W J,Li D X,Xu Y H,et al.Scavenger receptor B protects shrimp from bacteria by enhancing phagocytosis and regulating expression of antimicrobial peptides[J].Developmental and comparative immunology,2015,51(1):10-21.
[17]Yang D,Han Y,Chen L,et al.Scavenger receptor class B type I (SR-BI) in Ruditapes philippinarum:A versatile receptor with multiple functions[J]. Fish & shellfish immunology,2019,88:328-334.
[18]Zhang X,Dai L,Wu Z,et al.Molecular cloning,mRNA expression,and characterization of heat shock protein 10 gene from humphead snapper Lutjanus sanguineus[J].Marine genomics,2011,4(3):143-150.
[19]Silverstein R L,Febbraio M.CD36,a scavenger receptor involved in immunity,metabolism,angiogenesis,and behavior[J].Science signaling,2009,2(72):re3.
[20]Rosano G L,Ceccarelli E A.Recombinant protein expression in Escherichia coli:advances and challenges[J].Frontiers in microbiology,2014,05:172.
[21]黃瑜,牛金中,汤菊芬,等.尼罗罗非鱼NCCRP-1基因的原核表达及条件优化[J].基因组学与应用生物学,2018,37(09):3753-3758.
[22]Donovan R S,Robinson C W,Glick B R., et al.Optimizing the expression of a monoclonal antibody fragment under the transcriptional control of the Escherichia coli lac promoter[J].Canadian journal of microbiology,2000,46(6):532-541.
[23]Meng Z,Zhang X Y,Guo J,et al.Scavenger receptor in fish is alipopolysaccharide recognition molecule involved in negative regulation of NF-κB activation by competing with TNF receptor-associated factor 2 recruitment into the TNF-α signaling pathway[J].Journal of immunology,2012,189(8):4024-4439.
[24]杨晰朦,陈香梅.结核分枝杆菌ESAT6-CFP10融合蛋白的原核表达及其表达条件的优化[J].中国生物制品学杂志,2020,33(03):286-292.
(责编:张宏民)
基金项目:广东省自然科学基金项目(2016A030313748);大学生创新创业训练计划(CXXL2019139);南方海洋科学与工程广东实验室(湛江)自主项目(ZJW-2019-06)。
作者简介:王一帆(1996—),男,河北人,硕士生,研究方向:水产动物病害防治。 *通讯作者? 收稿日期:2020-12-24
- 公路工程结构物混凝土外观质量控制研究
- 提高民航通信导航监视系统可靠性的研究
- 刍议水利水电工程中碾压混凝土大坝的施工技术
- 基于Excel的生产计划表自动分析
- 探究苏里格气田排水采气技术进展及对策
- 侧扫声呐声波掠射角对海底管道检测的影响
- 机电工程技术在智能电网建设中的应用
- 汽车离合器的认知与检修
- 核电厂高压安注泵水力特性对全流量试验的影响分析
- 浅谈两种机械化便桥在施工中的应用
- 关于电网规划现状及展望的探索
- 武安市矿山镇铁矿冒落塌陷分析与预测
- 煤矿回采巷道矿山压力控制与支护分析
- 运行核电机组安全改进措施探讨
- 某核电站给水泵泵轴腐蚀原因分析及处理
- 聚合物泥浆在复杂地质钻孔桩施工中的应用
- 智能变电站继电保护系统所面临的若干问题
- 基于旅游地生命周期理论影响下的张掖丹霞旅游景区保护与开发
- 浅谈国际电子政务发展趋势及经验借鉴
- 宜昌文化创意产业存在的问题及高质量发展的对策研究
- 中国与西亚国家的电子商务对比研究
- 共享经济发展的现状及其思考
- 化工企业财务风险及其影响因素分析
- 会计信息化对企业财务管理的影响及对策
- 浅析我国上市公司财务舞弊
- faa
- fab
- fable
- fabler
- fablers
- fables
- fabling
- fabric
- fabricate
- fabricated
- fabricates
- fabricating
- fabrication
- fabrication/fab/fabricator/factory
- fabricative
- fabricator
- fabricators
- fabrics
- fabulous
- fabulously
- fabulousness
- fabulousnesses
- facade
- facaded
- facade, façade
- 墙有耳
- 墙有耳,伏寇在侧。
- 墙有风
- 墙根
- 墙根底下避雨——淋不上
- 墙皮
- 墙破麻雀多,人穷灾难多
- 墙纸
- 墙缝
- 墙缝的蝎子蜇人不显身
- 墙缝里的蚂蚁
- 墙缝里的蛇咬人
- 墙缝里的蛇咬人——出嘴不出身
- 墙缝里的蝎子
- 墙缝里的蝎子——蜇人不显身
- 墙脆脆
- 墙脚
- 墙花柳
- 墙花路柳
- 墙花路草
- 墙茨
- 墙葬
- 墙衣
- 墙角
- 墙角开口——斜(邪)门儿