田佳 吴楠 解诗雨 黄志伟 原尚奇

摘 ? ?要:为探究土壤环境中抗生素抗性基因宿主微生物的多样性,本研究以天津地区的农田土壤为研究对象,利用高通量测序技术对土样中大环内酯类抗性基因ermF的宿主细菌在不同分类水平的多样性进行了评估。土样中宿主细菌种类覆盖9个门,39个科,42个属。4个土样之间所共有的OTUs只占总OTUs数目的0.5%,说明样品间宿主细菌群落的差异性显著,可能主要与采样地点不同有关。在所有土样中,ermF基因的优势宿主细菌在门的水平上均为拟杆菌门(Bacteroidetes,相对丰度50.3%~87.7%);同时检测到少量的变形菌门(Proteobacteria),放线菌门(Actinobacteria)和厚壁菌门(Firmicutes),占比均小于4.2%。土样中ermF优势宿主細菌在科水平上都为拟杆菌科(Bacteroidaceae,相对丰度50.3%~87.5%),在属水平上都为拟杆菌属(Bacteroides,相对丰度50.3%~87.5%),其中相对丰度大于0.1%的ermF的宿主菌属有14种。
关键词:抗生素抗性基因;土壤;高通量;多样性
中图分类号:S963.91 ? ? ? ? ? ?文献标识码:A ? ? ? ? DOI 编码:10.3969/j.issn.1006-6500.2019.11.011
Abstract: To explore the host bacteria diversity of antibiotic resistance genes in soils, the high-throughput sequencing technique were used to evaluate the diversity of host bacteria of ermF, a macrolide resistance gene, at different taxonomic levels in farmland soils in Tianjin. The bacterial species in the samples covered 9 phyla, 39 families and 42 genera. The OTUs shared by the four soil samples only accounted for 0.5% of the total number of OTUs, which indicated that the difference of host bacterial community among the samples was significant, which might be related to the different sampling sites. At phylum level, the dominant host bacteria of ermF gene were Bacteroidetes (relative abundance 50.3%~87.7%) in all soil samples; a small number of Proteobacteria, Actinobacteria and Firmicutes were also detected, accounting for less than 4.2%. The dominant host bacteria of ermF were Bacteroidaceae (relative abundance 50.3%~87.5%) at family level, and Bacteroides(relative abundance 50.3%~87.5%) at genus level. There were 14 genera of ermF host bacteria with relative abundances > 0.1%.
Key words: antibiotic resistance genes; soil; high-throughput sequencing; diversity
抗生素在禽畜集约化养殖等方面的广泛应用,带动了畜禽养殖业的飞速发展。但抗生素的过量使用,也带来了一系列环境问题。长期对土壤施用残留有抗生素的畜禽粪便,除了会造成土壤中抗生素的积累,还会加快抗生素耐药性的传播。抗生素抗性基因(antibiotic resistance genes, ARGs),作为一种新型的环境污染物,因其能在环境中可持久性的残留且危害特别严重而受到人们的广泛关注[1]。ARGs借助于基因横向转移机制,可以在土壤及各个环境介质中迁移转化,对人类健康和生态环境带来一定风险。
大环内酯类抗生素是由链霉菌产生的一类弱碱性抗生素,多为碱性亲脂性化合物,能不可逆地结合到细菌核糖体亚基上,通过阻断转肽作用及位移,选择性抑制蛋白质合成。ermF基因是一种常见的大环内酯类抗性基因,在污泥、畜禽粪便等环境介质中都有检出,且具有较高丰度[2-3]。但目前针对环境介质中ermF基因宿主细菌多样性的研究还较少[4]。本研究以天津地区的农田土壤作为研究对象,利用高通量测序技术对土壤中ermF基因宿主细菌多样性进行系统分析,有助于评估环境中ARGs所带来的生态及健康风险。
1 材料和方法
1.1 土样采集
土壤样品为2016年3月到5月期间采自天津的蓟州(JZ),大港(DG),静海(JH)和宝坻(BD)地区的农田土样。每个样点采集表层土壤0~10 cm。
1.2 高通量测序
ermF基因的高通量测序在北京奥维森公司利用Illumina MiSeq平台进行。土壤DNA提取采用Power Soil DNA Isolation Kit试剂盒 (MoBio, USA)。表1列出了ermF基因扩增所用引物和退火温度[5]。
1.3 生物信息学和统计分析
首先,对原始测序数据进行过滤处理,过滤reads尾部质量值20以下的碱基,设置50 bp的窗口,如果窗口内的平均质量值低于20,从窗口开始截去后端碱基,过滤质控后50 bp以下的reads;然后根据reads的overlap关系将成对的序列拼接成一条序列,最小overlap长度为10 bp,拼接允许的最大错配比率为0.1。根据序列首尾两端的barcode和引物区分样品,并调整序列方向,barcode允许的错配数为0。用Usearch软件去除嵌合体,通过Mothur去掉长度较小的tags[6]。在0.97相似度下利用UCLUST软件将拼接过滤后的序列聚类用于物种分类的OTUs(operational taxonomic units)。样品中每个OTU的代表性序列,通过BLAST工具与NCBI NR数据库进行比对,得到对应物种分类信息。
2 结果与分析
2.1 宿主细菌群落多样性分析
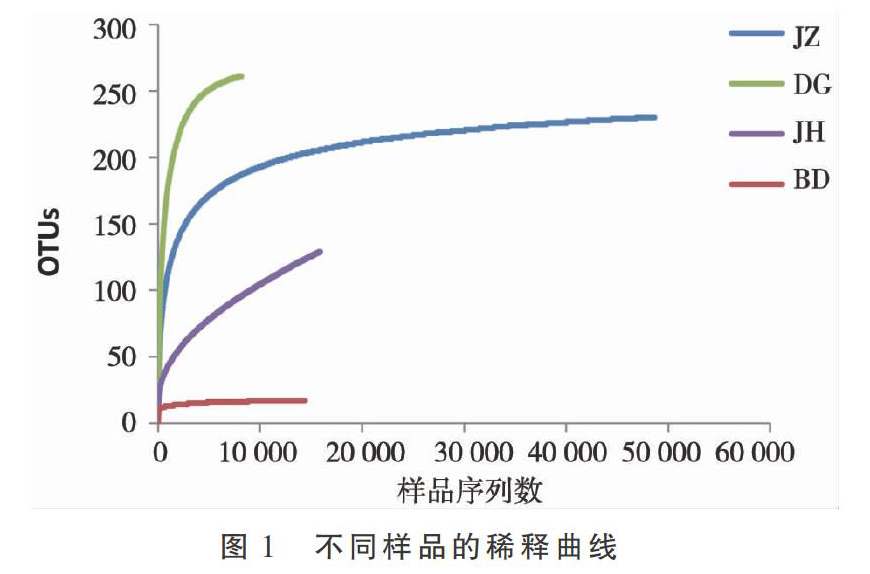
4个土样进行测序后共获得285 234条高质量序列,优质序列长度集中在 240~300 bp。测序覆盖深度(Coverage指数)均在0.99以上,说明测序结果代表了样本中微生物的真实情况。各样品的稀释曲线能在一定程度上反映不同样品菌群多样性。由图1可以看出, JZ、DG和BD的稀释曲线已经趋于平坦,说明测序数据足以覆盖所有的微生物。JH的稀释曲线虽然趋于平缓,但仍未达到平台期,说明测序量仍有部分的宿主细菌群不能够被覆盖,如果增大测序量,仍然有可能发现新的OTUs[6]。
Shannon指数指示了样品的细菌多样性的丰富程度,数值越高,表明细菌群落的物种多样性越高,分布越均匀。JZ土样的Shannon指数为3.87,远高于其他3个土样(DG:1.48;JH:1.79;BD:1.43)。这表明在4个土样中,JZ土样中的ermF基因宿主细菌群落的多样性程度最高。
2.2 OTUs分布维恩图
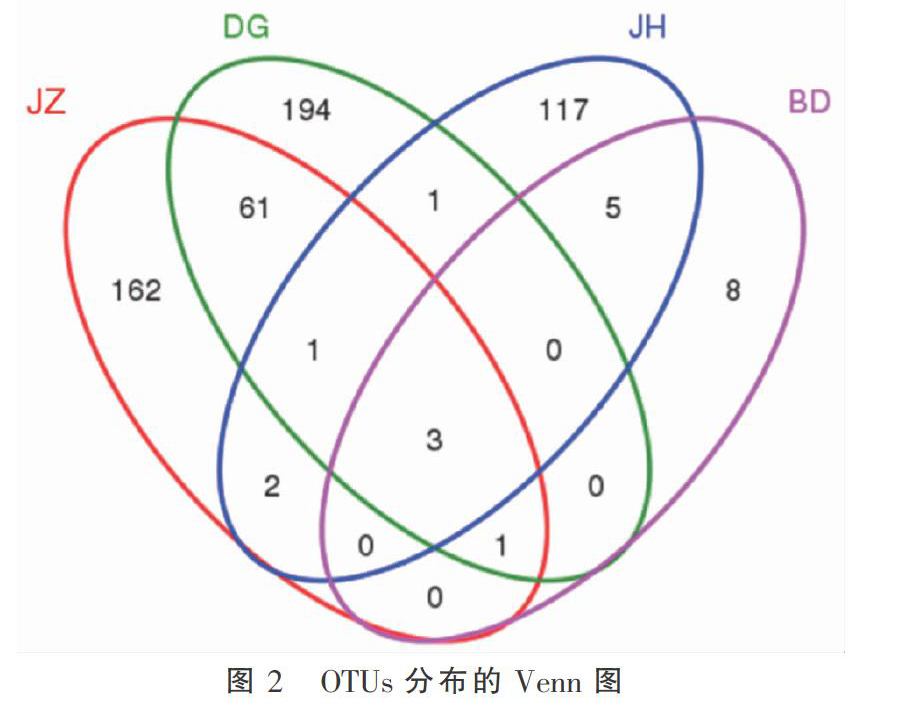
图2 直观地展现了4个土样中所共有和独有的OTUs 数目及重叠情况。4个土样中所共有的OTUs只有3个,占总OTUs数目(555个)的0.5%。除BD土样外,其余3个土样各自独有OTUs数目均超过100个。结果表明所测土样之间宿主细菌群落的差异性明显。
2.3 在门水平上的群落丰度分析
由图3可知,4个土样中共分离得到9个微生物菌门,其中相对丰度小于0.1%的菌门归为其他类。在门类水平上,所有土样中ermF的宿主优势菌都为拟杆菌门(Bacteroidetes),其中DG,JH和BD土样中拟杆菌门占75.7%~87.7%,而在JZ土样中比例较低,约占50.3%。此外,ermF的优势宿主菌还有变形菌门(Proteobacteria),在JZ和JH土样中分别占4.1%和3.9%,而在其余土样中占比都小于0.5%。JZ土样中还检测到了少量的放线菌门(Actinobacteria)和厚壁菌门(Firmicutes),相对丰度分别为0.7%和0.2%。
2.4 在科水平上的群落丰度分析
土样中共检测得到39个微生物菌科,其中相对丰度小于0.1%的菌科归为其他类。由图4可知,所有土样中ermF的宿主优势菌在科水平上都为拟杆菌科(Bacteroidaceae),相对丰度为50.3%~87.5%。JZ土样中的多囊粘菌科(Polyangiaceae)、葉杆菌科(Phyllobacteriaceae)和丙酸杆菌科(Propionibacteriaceae)的相对丰度较高,分别为1.6%,0.9%和0.4%。在JH土样中也检测到了较高丰度的叶杆菌科,相对丰度为3.6%。在BD土样中检测到较高丰度的紫单胞菌科(Porphyromonadaceae),相对丰度为1.1%。其他微生物菌科在4个土样中所占比例极少,均低于0.4%。
2.5 在属水平上的群落丰度分析
土样中共分离得到42个微生物菌属。图5显示了在属水平上的ermF基因宿主细菌群落相对丰度热图,其中相对丰度小于0.1%的菌属归为其他类。在JZ样品中,ermF基因的宿主细菌中占比最多的3种菌属分别为拟杆菌属(Bacteroides,相对丰度50.3%),黏细菌属(Chondromyces,相对丰度1.6%)和根瘤菌属(Mesorhizobium,0.9%)。在其他3个土样中,ermF基因的宿主细菌中占比最多的同样为拟杆菌属(相对丰度75.7%~87.5%)。在JH土样中检测到较高丰度的根瘤菌属(3.6%)。此外,巴氏杆菌属(Barnesiella)在所有土样中都被检测到,其中BD土样中的巴氏杆菌属的相对丰度最高(1.1%)。值得注意的是,许多宿主菌属只在JZ土样中检测到(相对丰度0.1%~0.4%),而没有出现在其他3个土样中,如原囊菌属(Archangium),固氮螺菌属(Azospirillum),克洛诺菌属(Cronobacter),鞘脂单胞菌属(Sphingomonas),葡萄球菌属(Staphylococcus)和硫针菌属(Sulfuritalea)。这表明JZ土样中的ermF的宿主细菌落多样性较高,这与之前的Shannon指数结果一致。
3 结论与讨论
本研究通过高通量测序技术,对天津地区的农田土壤中大环内酯类抗性基因ermF的宿主细菌在不同分类水平的多样性进行了分析。4个土样中ermF基因的宿主细菌种类覆盖9个门,39个科,42个属。4个土样之间所共有的OTUs只占总OTUs数目的0.5%,说明样品间宿主细菌群落的差异性明显,这可能主要与采样地点不同有关。在所有土样中,ermF基因的优势宿主细菌在门的水平上均为拟杆菌门(Bacteroidetes,相对丰度50.3%~87.7%);同时检测到少量的变形菌门(Proteobacteria),放线菌门(Actinobacteria)和厚壁菌门(Firmicutes),占比均小于4.2%。土样中ermF优势宿主细菌在科水平上都为拟杆菌科(Bacteroidaceae,相对丰度50.3%~87.5%),在属水平上均为拟杆菌属(Bacteroides,相对丰度50.3%~87.5%)。这与前人研究结果一致,Eitel等[7]曾报道在拟杆菌的菌株中检测到高丰度的ermF基因。Johnsen等[8]发现大环内酯类抗性基因(erm)通常存在于拟杆菌属中。在42个ermF宿主菌属中,相对丰度大于0.1%的菌属有14个。Zhang 等[9]报道在污泥堆肥体系中,ermF基因的潜在宿主菌属高达24个,其中如葡萄球菌属(Staphylococcus)和假单胞菌属(Pseudomonas),在本研究中也被检测到。和JZ土样相比,其他土样中的拟杆菌属的占比增加,造成这一现象的原因可能与采样地点不同,土壤的基本理化性质差异有关。例如有研究表明拟杆菌属在富营养环境中能够更好地存活[10]。环境因子对于ermF宿主细菌群落结构多样性的影响还需进一步研究。本文结果为探索抗生素抗性基因宿主细菌的多样性提供了科研依据与基础。
参考文献:
[1]WU N, QIAO M, ZHANG B, et al. Abundance and diversity of tetracycline resistance genes in soils adjacent to representative swine feedlots in China[J]. Environmental science & technology, 2010, 44: 6933-9.
[2]韦蓓, 黄福义, 李虎, 等. 污泥堆肥过程中磺胺类和大环内酯类抗性基因的残留[J]. 应用与环境生物学报, 2014, 20(3): 395-400.
[3]郑宁国, 黄南, 王卫卫, 等.高温堆肥过程对猪粪来源抗生素抗性基因的影响[J].环境科学,2016, 37(5): 1986-1992.
[4]CHUNG W O, WERCKENTHIN C, SCHWARZ S, et al. Host range of the ermF rRNA methylase gene in bacteria of human and animal origin[J]. Journal of antimicrobial chemotherapy, 1999, 43(1): 5-14.
[5]CHEN J, YU Z, MICHEL F C, et al. Development and aplication of real-time PCR assays for quantification of erm genes conferring resistance to macrolides-lincosamides-streptogramin B in livestock manure and manure management systems[J]. Applied and environmental microbiology, 2007, 73(14): 4407-4416.
[6]周美丽, 王立志, 闫天海, 等. 林麝粪便细菌多样性研究[J]. 中国農业大学学报, 2016, 21(2): 100-106.
[7]EITEL Z, SOKI J, URBAN E, et al. The prevalence of antibiotic resistance genes in Bacteroides fragilis group strains isolated in different European countries[J]. Anaerobe, 2013, 21: 43-49.
[8]JOHNEN B O, HANDAL N, MEISAL R, et al. erm gene distribution among Norwegian Bacteroides isolates and evaluation of phenotypic tests to detect inducible clindamycin resistance in Bacteroides species[J]. Anaerobe, 2017, 47: 226-232.
[9]ZHANG J, CHEN M, SUI Q, et al. Impacts of addition of natural zeolite or a nitrification inhibitor on antibiotic resistance genes ?during ?sludge composting[J]. Water research, 2016, 91: 339-349.
[10]WANG M, LIU P, XIONG W, et al. Fate of potential indicator antimicrobial resistance genes (ARGs) and bacterial community diversity in simulated manure-soil microcosms[J]. Ecotoxicology and environmental safety, 2018, 147: 817-823.
- 中小企业网络招聘存在的问题及对策
- 经济转型中民营企业人力资源管理方式的转变
- 大数据驱动的人力资源绩效管理探究创新
- 5G:商业银行数字化转型的重要引擎
- 金融科技在厅堂风险防控中的应用
- 金融科技如何影响金融监管和金融消费权益保护
- 探索银行数字化转型中台战略
- 金融机构应强化销售环节的适当性义务
- 民间借贷的律师费辨析
- 金融监管与金融审判的冲突与合作
- 债券通业务的发展现状及未来展望
- 投资者适当性管理的提升对策
- 客户信用分析的逻辑
- “存量时代”的不良资产处置
- BigTech涉足金融基础设施建设的逻辑与建议
- 股权信托受托人的特别谨慎义务及其免除的合法性
- 从中国财富管理收益指数报告透视中国资管市场
- 基层农商行管理九诫
- 科技+生态赋能 打造对公业务“发动机”
- 经济改革大潮中的理论家们
- 2019年英国银行业发展述评
- 2019年美国银行业发展回顾与展望
- 变革中的英美银行业
- 推动农商银行高质量发展实现新跨越
- 慈善公益金融参与“一带一路”的策略
- nonfigurativenesses
- nonfilamentous
- nonfiler
- nonfilers
- nonfilter
- nonfilterable
- nonfilters
- nonfinal
- nonfinancial
- nonfinancially
- nonfinding
- nonfindings
- nonfinishing
- nonfinishings
- nonfinite
- nonfinitely
- nonfiniteness
- nonfinitenesses
- nonfinites
- nonfireproof
- nonfiscal
- nonfiscally
- nonfisherman
- nonfishermen
- nonfixation
- 醉人的蒸馏酒
- 醉人说梦
- 醉令
- 醉会
- 醉伴
- 醉侣
- 醉侯
- 醉倒
- 醉倒山公
- 醉倒玉山
- 醉倒马杓
- 醉兀
- 醉兀兀
- 醉八仙
- 醉公子
- 醉击珊瑚枝
- 醉卧
- 醉卧干日
- 醉卧翠馆花丛
- 醉卧酒垆侧
- 醉后不稳的步子
- 醉后东倒西歪的样子
- 醉后两眼模糊不清的样子
- 醉后使性子
- 醉后的体态