DNA甲基化及其在水产养殖动物中的应用研究进展
王昊泽+王秀利

摘 要:DNA甲基化是真核生物基因表达调控的重要的一种表观遗传机制。近年来,随着对水产养殖动物各领域研究的不断深入,DNA甲基化在水产养种动物遗传育种的领域引起了广泛的关注,取得了一些进展。本文简要介绍了DNA甲基化和其检测技术,并从鱼类、贝类、棘皮动物三个方面分别介绍了该技术在这三类动物上的研究进展,为今后水产养殖动物的表观遗传学研究提供参考。
关键词:DNA甲基化;水产动物;研究进展
DNA甲基化(DNA methylation)又称DNA的共价修饰,是重要的表观遗传(epigenetics inheritance)现象之一。表观遗传是指在不改变DNA序列的前提下,产生可遗传的核酸序列修饰,并得到不同的表现型。其中DNA甲基化对基因的表达有重要的调控作用,例如:DNA甲基化能关闭某些基因的活性,去DNA甲基化则诱导了基因的重新活化和表达;DNA甲基化还能引起染色质结构、DNA构象、DNA稳定性及DNA与蛋白质相互作用方式的改变,从而控制基因表达[1]。
有研究证实,在发生人癌变的细胞内肿瘤细胞中癌基因DNA甲基化水平降低;抑癌基因的甲基化水平高;加之错配修复基因表达缺陷的原因不仅有基因突变,其基因启动区也呈高甲基化水平[2]。在动植物研究方面越来越多的实验表明,DNA甲基化在动物、植物的生长发育中起着举足轻重的作用。通过对植物和哺乳动物DNA甲基化的比较分析,有助于我们了解RNA引导的DNA甲基化机制。在哺乳动物中,全基因组DNA甲基化和去甲基化发生在配子形成和早期胚胎发育[3]。这些过程可能涉及多個DNA甲基化或去甲基化机制。
1 DNA甲基化与检测技术
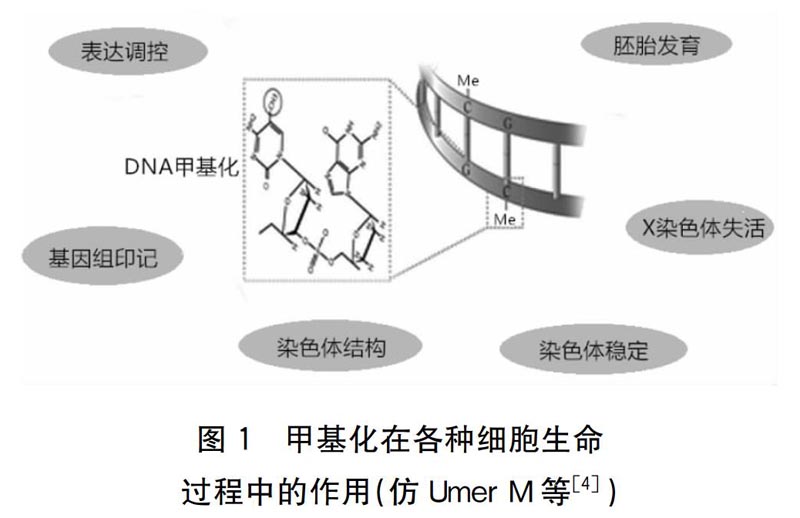
表观遗传学中研究最清楚、最重要的修饰形式就是DNA甲基化。它在细胞发育和运行正常功能过程中起着很多关键作用,包括基因表达调控、胚胎发育、基因组印记和染色体稳定性等[4] (图1)。
DNA甲基化的含义即在甲基转移酶的催化下,DNA的CG两个核苷酸的胞嘧啶被选择性地添加甲基,形成5-甲基胞嘧啶,常见于基因的5'-CG-3'序列。大多数脊椎动物基因组DNA都有少量的甲基化胞嘧啶,主要集中在基因5'端的非编码区,并成簇存在。
DNA甲基化具有自我永续结构(图2),其特点为甲基化等位基因复制产生的半甲基化子链,它通过组成活性甲基化酶恢复其甲基化活性状态。
DNA甲基化的检测技术大致可以分为两类:特异位点的甲基化检测和全基因组的甲基化分析,后者也称为甲基化图谱分析(methylation profiling)。特异位点的甲基化检测有以下几种重要方法:(1)甲基化特异性PCR(MS-PCR);(2)亚硫酸氢盐处理+测序;(3)联合亚硫酸氢钠的限制性内切酶分析法(COBRA);(4)荧光定量法(Methylight);(5)甲基化敏感性高分辨率熔解曲线分析;(6)焦磷酸测序。基于芯片的甲基化图谱分析一般使用的芯片有安捷伦的Human CpG Island Microarray Kit,Illumina的HumanMethylation27 DNA analysis BeadChip,Roche NimbleGen的Human DNA Meth 2.1M Deluxe Promoter Array和Affymetrix的seven-array GeneChip Human Tiling 2.0R Array Set。[5]还有利用将传统的甲基化工具(如DNA的亚硫酸氢盐转化)与目标基因组捕获技术和高通量测序相结合,绘制出甲基化图谱的研究方法[6]。此外MassARRAY EpiTYPERTMDNA 甲基化分析技术结合了碱基特异性酶切反应和 MALDI-TOF 检测原理,可实现多重CpG的分析检测,这种方法被称为飞行质谱[7]。
2 甲基化在养殖鱼类上的应用
种群的性别决定机制决定其性别比例,是其稳定性的一个重要的种群参数[8]。鱼是变温(冷血)动物,其胚胎发育受物理环境的影响较大。对于某些鱼类物种,有越来越多的证据表明,温度影响性别决定[9]。有13科60多种不同的鱼类属于温度依赖型性别决定(Temperature-dependent sex determination,TSD)[10-11]。TSD是在基因型性别决定(Genotypic sex determination,GSD)的基础上,通过温度效应(Temperature effects,TE)实现的[12]。温度对鱼类性别具体的影响机制大体可分为这几个方面:主要调控性别的基因[13]、睾丸决定基因[14]、卵巢决定基因[15]、皮质醇介导调控途径[16]和表观遗传调控途径[17]。
在这里我们着重介绍在温度调控下的DNA甲基化对鱼类性别的影响。
温度的变化对欧洲海鲈鱼性别比例有较大影响。欧洲海鲈鱼为雌雄异体,一种典型的温度依赖型性别决定(TSD)和基因型性别决定(GSD)结合决定的[8]。虽然明确了欧洲海鲈鱼的性别决定机制为TSD和GSD结合型,但性别比例和温度的相互关系及其机制仍然令人费解。科学家Laia Navarro-Martín等(2011)[18]利用欧洲海鲈鱼为实验样本,验证他们提出的假设:“存在一个表观遗传机制激活温度,可能导致性腺发育的启动子的DNA甲基化水平不同,反过来又会影响基因的表达–雌激素的合成,因此性别比例受到影响。实验结果表明:(1)欧洲鲈鱼性腺发育基因(CYP19A)启动子的甲基化水平受性别和在生命早期经历的温度的影响;(2)欧洲海鲈鱼在早期生活中经历的高温能够使群体雄性增多,其原因是高温降低了CYP19A mRNA在雌性生殖腺的表达水平;(3)外源性雌激素的摄入并没有改变海鲈鱼的CYP19A启动子甲基化水平;(4)在海鲈CYP19A启动子特异性CpG序列的甲基化水平由性别和温度决定。对欧洲海鲈鱼的研究还发现温度与多个性别决定基因的遗传贡献是相同的[19-20]。
半滑舌鳎(Cynoglossus semilaevis)是我国渤、黄海的地方性名贵鱼种,由于过度捕捞和生境变化,资源量严重衰竭,加之独特的生殖习性(雌雄型体差异悬殊和性比变化幅度较大),其种质资源现状及其可持续利用令人堪忧[21]。因为半滑舌鳎雌雄个体生长差异悬殊,由于性逆转而造成的其群体中雌性比例过低大大制约了养殖效率[22]。在养殖技术研究过程中,Chen等(2012)[23]发现,利用人工雌核发育创制的超雌个体可能只能存活到肌节期和出膜前期。 研究人员Shad CW等(2014)[24]通过大量家系性别比例检测以及 DNA甲基化分析发现伪雄鱼的染色体的DNA甲基化修饰模式与其后代一致。说明DNA甲基化模式被烙印在基因组中,在新世代的发展过程中没有被清除和重置,所以它们出现在后代中。李启业等人(2013)[25]通过对半滑舌鳎的性腺进行全基因组甲基化比较分析,发现基因型雌鱼逆转为伪雄鱼的过程中,性别决定通路中很多基因发生了甲基化重塑,说明用于GSD过程的遗传因子通过表观调控同样可以用于实现ESD过程。此外,形成伪雄鱼中发生的DNA甲基化改变在它们的雌性后代中维持了下来,而这些后代雌鱼在没有高温诱导的条件下也能自然转变为伪雄鱼。还通过对比较不同性腺的转录组,发现Z染色体上有一个甲基化位点特别富集的区域在伪雄鱼的精巢中发生基因表达的上调,使表达水平跟正常雄鱼的精巢持平,说明这个区域携带了一些对精巢来说功能重要且剂量敏感的基因。相反,W染色体上的雌性特异基因通过甲基化调节在伪雄鱼中被抑制。
孙瑞健等(2012)[26]发现,大黄鱼的肝脏和肌肉脂肪含量、肝脏指数(HSI)以及内脏指数(VSI)受到了饲料脂肪水平的显著影响(P<0.05),在2次/天投喂时,肝脏和肌肉脂肪含量、HSI以及VSI随饲料脂肪水平的提高而显著增加(P<0.05);Liao K等(2015)[27]发现,膳食脂肪源通过对大黄鱼肝部线粒体DNA甲基化的影响,从而转变大黄鱼的线粒体功能,影响其生长发育。
Jun Xiao等(2012)[28]对红鲫异源四倍体鲫鲤杂交种(Carassius auratus red var.) 和鲤鱼(Cyprinus carpio)进行了DNA甲基化分析。发现其甲基化修饰水平增加量基于亲鱼的甲基化基数;一些调控基因例如Sox9a、 4nAT等发生了数量上的变化。这些结果的间接证据表明,DNA甲基化可能与异源四倍体鲫鲤的表型变异的基因表达相关。
3 甲基化在养殖贝类中的应用
由中国科学院海洋研究所和深圳华大基因研究院领导的一个国际研究小组完成了对太平洋牡蛎(Crassostrea gigas)的测序、组装与分析,这是第一个测序的软体动物基因组,将有助于填补我们对于种类丰富而较少研究的软体动物家族的了解空白[29]。
Wang等(2014)[30]在太平洋牡蛎基因组测序的基础上,对模式软体动物的全基因组进行了DNA甲基化分析。他们分析了牡蛎中DNA甲基化的一般特点,用它与其他无脊椎动物已发表的甲基化组比较,并从其他物种提供的CpG甲基化演变中寻找新的见解。经比较后的结论认为,牡蛎基因组是一个编码完整的甲基化的工具;牡蛎基因组甲基化水平远高于昆虫,但接近其他无脊椎动物;CpG岛甲基化的优先目标基因起源于真核生物的“祖先”基因而不是古老的基因等。经推测,在真核生物的“祖先”基因中出现新的基因,一起对这些基因的CpG岛甲基化形成更复杂的调控机制,这一点在真核生物的起源和发展方面是很重要的。
利用牡蛎的表观遗传学特征研究了无脊椎动物中相关启动子DNA的甲基化。科学家Rivière等(2014)[31]认为,在无脊椎动物的DNA甲基化与转录、选择性剪切和基因组进化有关,但有关记录和描述在冠轮动物门中少之又少。他认为目前的全基因组研究带来了无脊椎动物的甲基化组信息的静态图片;研究应该从高通量技术破译这种DNA甲基化组的动力学及相关功能的影响机制。作为一种模式生物,其基因组广泛用于研究貝类生物分子生物学机制。从目前已有的研究结果看,功能相关的特定基因甲基化特征,能更好地了解长牡蛎DNA的甲基化作用,可以大大提高我们对无脊椎动物这种表观遗传标记的理解。
Olson等(2013)[32]利用全基因组测序详细描述了雄配子细胞全基因组DNA甲基化谱。他们选用了一个从Thorndyke湾收集的雄性成年牡蛎,在实验室模拟自然环境培养六周,取雄配子体组织,用消毒的海水清洗配子后,离心,并立即放在干冰内,然后放在-80℃直到进一步处理雄性配子。提取DNA,建立DNA文库。最终生成为了表征的雄配子体组织中甲基化分布。分析结果表明:CpG DNA甲基化在生物中无处不在,在无脊椎动物和脊椎动物中的甲基化水平与方式有很大的不同,大多数无脊椎动物基因组表现出散布区域的甲基化和非甲基化DNA;有一个相关性高的甲基化实例发生在牡蛎基因组中,但没有明确的证据表明甲基化和功能之间的联系;基于RNA-Seq数据有可能是DNA甲基化状态与太平洋牡蛎基因表达之间的关联;有更高比例的CpG岛甲基化的启动子是与基因的高表达有关的。
扇贝(Pectinidae)是我国重要的贝类养殖品种,具有很高的营养价值[33]。Sun等(2014)[34]利用MSAP技术,对扇贝的五个组织(鳃、肾、卵巢、平滑肌和横纹肌)进行了基因组范围的检测。在同一养殖网箱,选用两龄的栉孔扇贝(Chlamys farreri)个体,共收集6个雌性个体,进行甲基化分析,发现在不同的组织中检测到甲基化水平为20.9%~21.7%,并没有显著的差异。栉孔扇贝不同组织中的甲基化变异相对较小(0.74%),而其他生物的变异较大(1.54%~3.81%)(玉米的甲基化水平20.24%~21.78%[35]、鸡的甲基化水平为26.1%~29.4%[36]、猪的甲基化水平为50.18%~53.99%[37]),说明在扇贝组织之间甲基化水平可能不发散。MSAP数据结果表明,有64%到71%的未甲基化的epiloci型,53%到57%的hmecgMECG epiloci型和40%到47%的hmeccg epiloci型。DNA甲基化的多样性是不可能受到环境影响的,甲基化差异可能与栉孔扇贝基因组杂合度高有关;甲基化差异可能是由于DNA去甲基化;DNA去甲基化对甲基化模式的塑造起着举足轻重的作用,其潜在的机制仍不完全清楚。
4 甲基化在棘皮動物(Echinoderm animal)上的应用
国内仿刺参(Apostichopus japonicus)养殖规模不断扩大 ,养殖方式多种多样。然而养殖的过速发展和不规范运作也造成了如生态环境恶化、病害等诸多问题[38]。仿刺参有夏眠的生活习性。当水温达20℃时,刺参就会转移到深海的岩礁缝隙中或潜藏于石底,不吃不动,整个身体收缩变硬如刺球。科学家针对仿刺参夏眠的状态,基于荧光标记甲基化敏感扩增多态性(F-MSAP),对刺参的四种组织进行了甲基化水平分析。Zhao等 (2014)[39]利用刺参夏眠三个不同阶段的刺参组织,进行甲基敏感扩增片段多态性分析(F-MSAP)。使用18对选择性引物,共扩增出26 963个片段,其中四个仿刺参组织中有9 112个甲基化片段。结果表明,刺参DNA甲基化水平平均33.79%。DNA甲基化的发生率在非夏眠阶段的组织类型不同:小肠(30.16%)、呼吸树(27.61%)、肌肉(27.94%)和体壁(56.25%)。研究结果表明,DNA甲基化伴随着刺参深度夏眠,可能在深度夏眠期间是调节全局转录抑制的一个重要的角色。进一步分析表明,DNA修饰位点主要集中在肠和呼吸树组织,在深度夏眠阶段全甲基化比半甲基化水平显著增加。
海胆(Sea urchin)是一种低等的海洋无脊椎动物,属于棘皮动物门海胆纲。海胆黄为名贵的海产珍品,具有极高的营养价值和药用价值[40]。James等(1979)[41]在研究海胆精子和胚胎5-甲基胞嘧啶的含量时发现,通过分析高压色谱柱分离的aminex-10甲酸水解产物,确定海胆精子和胚胎早期卵裂阶段的胞嘧啶和5-甲基胞嘧啶的DNA之间的比例。他们发现在胚胎细胞核不同的发展阶段,在DNA的甲基化水平方面找不到任何可测量的变化。还发现海胆的精子和胚胎细胞核的5-甲基胞嘧啶的甲基化水平含量之间没有差异。
5 结语
目前,中国水产业发展形势严峻。一方面,大量的环境变迁与生境改变,致使因环境恶化导致许多水产生物资源量严重下降。另一方面,由于养殖业不健康的快速发展,致使病害频发,种质资源恶化等。而DNA甲基化作为一种相对稳定的表观遗传修饰,动态地调节着细胞、生物体的生命过程,将为水产养殖动物的表观遗传学应用于种质改良带来广阔的前景。
参考文献:
[1] Moore L D,Le T,Fan G.DNA Methylation and Its Basic Function[J].Neuropsychopharmacology Official Publication of the American College of Neuropsychopharmacology,2013,38(1):23-38
[2] 赵迎社,付步银.DNA甲基化在肿瘤形成中的作用(综述)[J].暨南大学学报:自然科学与医学版,2001,22(4):54-57
[3] He X J,Chen T,Zhu J K.Regulation and function of DNA methylation in plants and animals [J].Cell Research,2011,21(3):442-65
[4] Umer M,Herceg Z.Deciphering the epigenetic code: an overview of DNA methylation analysis methods [J].Antioxidants & Redox Signaling,2013,18(15):1972-86
[5] 孙贝娜.DNA甲基化检测方法的研究进展[J].生命科学仪器,2009,7(4):11-14
[6] Rodriguez B A,Frankhouser D,Murphy M,et al.Methods for high-throughput MethylCap-Seq data analysis [J].Bmc Genomics,2012,13(Suppl 6):387-390
[7] Singhal N,Kumar M,Kanaujia P K,et al.MALDI-TOF mass spectrometry: an emerging technology for microbial identification and diagnosis [J].Frontiers in Microbiology,2015,6:791
[8] Valenzuela N,Lance V.Temperature-dependent sex determination in vertebrates[M].Washington: Smithsonian Book .2004
[9] Conover D O,Kynard B E.Environmental sex determination: interaction of temperature and genotype in a fish [J].Science,1981,213(4507):577-9
[10] Ospina-Alvarez N,Piferrer F.Temperature-dependent sex determination in fish revisited: prevalence,a single sex ratio response pattern,and possible effects of climate change [J].Plos One,2008,3(7):e2837
[11] Baroiller J F,D'Cotta H,Saillant E.Environmental Effects on Fish Sex Determination and Differentiation [J].Sexual Development Genetics Molecular Biology Evolution Endocrinology Embryology & Pathology of Sex Determination & Differentiation,2009,3(2-3):118-35
[12] Valenzuela N,Adams D C,Janzen F J.Pattern does not equal process: exactly when is sex environmentally determined [J].American Naturalist,2003,161(4):676-83
[13] Devlin R H,Nagahama Y,Devlin R H,et al.Sex determination and sex differentiation in fish: an overview of genetic,physiological,and environmental influences [J].Aquaculture,2002,208(3-4):191-364
[14] Fergusonsmith M.The evolution of sex chromosomes and sex determination in vertebrates and the key role of DMRT1 [J].Sexual Development Genetics Molecular Biology Evolution Endocrinology Embryology & Pathology of Sex Determination & Differentiation,2007,1(1):2-11
[15] Chiang E F,Yan Y L,Guiguen Y,et al.Two Cyp19 (P450 aromatase) genes on duplicated zebrafish chromosomes are expressed in ovary or brain [J].Molecular Biology & Evolution,2001,18(4):542-550
[16] Hurk R V D,Oordt P G W J V.Effects of natural androgens and corticosteroids on gonad differentiation in the rainbow trout,Salmo gairdneri [J].General & Comparative Endocrinology,1985,57(2):216-222
[17] Gorelick R.Evolution of dioecy and sex chromosomes via methylation driving Muller's ratchet [J].Biological Journal of the Linnean Society,2003,80(2):353–368.
[18] Navarro-Martín L,Vi as J,Ribas L,et al.DNA methylation of the gonadal aromatase (cyp19a) promoter is involved in temperature-dependent sex ratio shifts in the European sea bass [J].Plos Genetics,2011,7(12):1176-1184
[19] Piferrer F,Blázquez M,Navarro L,et al.Genetic,endocrine,and environmental components of sex determination and differentiation in the European sea bass (Dicentrarchus labrax L) [J].General & Comparative Endocrinology,2005,142(1-2):102-10
[20] Vandeputte M,Dupont-Nivet M,Chavanne H,et al.A polygenic hypothesis for sex determination in the European sea bass Dicentrarchus labrax [J].Genetics,2007,176(2):1049-57
[21] 庄志猛.半滑舌鳎早期发育生物学与种质资源研究[D].中国海洋大学,2006.
[22] 宋超,蒋丽,王景伟,等.半滑舌鳎性逆转的遗传特性研究[J].生物技术通报,2015(3):207-212.
[23] Chen S L,Ji X S,Shao C W,et al.Induction of Mitogynogenetic Diploids and Identification of WW Super-female Using Sex-Specific SSR Markers in Half-Smooth Tongue Sole (Cynoglossus semilaevis) [J].Marine Biotechnology,2012,14(1):120-8
[24] Coyne K S,Sexton C,Kopp Z S,et al.Epigenetic modification and inheritance in sexual reversal of fish [J].Genome Research,2014,24(4):604-15
[25] 李启业.DNA甲基化对调控半滑舌鳎性别逆转的作用[D].华南理工大学,2013
[26] 孙瑞健.饲料蛋白质、脂肪水平与投喂频率对大黄鱼幼魚生长和饲料利用的影响[D].中国海洋大学,2012
[27] Kai L,Jing Y,Kangsen M,et al.Dietary Olive and Perilla Oils Affect Liver Mitochondrial DNA Methylation in Large Yellow Croakers [J].Journal of Nutrition,2015,145(11):2479-85
[28] Xiao J,Song C,Liu S,et al.DNA methylation analysis of allotetraploid hybrids of red crucian carp (Carassius auratus red var.) and common carp (Cyprinus carpio L.) [J].Plos One,2012,8(2):e56409-e56409
[29] Zhang G,Fang X,Guo X,et al.The oyster genome reveals stress adaptation and complexity of shell formation [J].Nature,2012,490(7418):49-54
[30] Wang X,Li Q,Lian J,et al.Genome-wide and single-base resolution DNA methylomes of the Pacific oyster Crassostrea gigas,provide insight into the evolution of invertebrate CpG methylation [J].Bmc Genomics,2014,15(1):1-12
[31] Rivière G.Epigenetic features in the oyster Crassostrea gigas suggestive of functionally relevant promoter DNA methylation in invertebrates.[J].Frontiers in Physiology,2014,5:129
[32] Olson C E,Roberts S B.Genome-wide profiling of DNA methylation and gene expression in Crassostrea gigas male gametes [J].Frontiers in Physiology,2013,5:224-224
[33] 王红.中国北方海域扇贝产品质量的比较研究[D].中国科学院研究生院(海洋研究所),2007.
[34] Sun Y,Hou R,Fu X,et al.Genome-Wide Analysis of DNA Methylation in Five Tissues of Zhikong Scallop,Chlamys farreri [J].Plos One,2014,9(1):e86232.
[35] Lu Y,Rong T,Cao M.Analysis of DNA methylation in different maize tissues [J].遗传学报,2008,35(1):41-48
[36] Xu Q,Zhang Y,Sun D,et al.Analysis on DNA methylation of various tissues in chicken [J].Animal Biotechnology,2007,18(4):231-41
[37] Yang C,Zhang M,Niu W,et al.Analysis of DNA methylation in various swine tissues [J].Plos One,2011,6(1):65-65
[38] 张春云,王印庚,荣小军,等.国内外仿刺参自然资源、养殖状况及存在问题[J].渔业科学进展,2004,25(3):89-97
[39] Zhao Y,Chen M,Storey K B,et al.DNA methylation levels analysis in four tissues of sea cucumber Apostichopus japonicus,based on fluorescence-labeled methylation-sensitive amplified polymorphism (F-MSAP) during aestivation [J].Comparative Biochemistry & Physiology Part B Biochemistry & Molecular Biology,2014,181:26-32
[40] 白日霞.馬粪海胆多糖的研究[D].大连理工大学,2010
[41] Jr P J,Swihart M,Taylor J H.Methylation of DNA in early development: 5-methyl cytosine content of DNA in sea urchin sperm and embryos [J].Nucleic Acids Research,1979,5(12):4855-61

