远东偏顶蛤核糖体DNA第二转录间隔区(ITS2)测序及分析
林杉杉+鲍相渤+刘忠颖+王超+杨慧花


摘要:利用通用引物成果扩增了远东偏顶蛤核糖体DNA第二转录间隔区序列,并对其进行了一致性比对和系统进化分析,结果表明:远东偏顶蛤ITS2序列全长共245 bp,四个个体的序列无差异。与其他物种相比,与远东偏顶蛤一致性最高的物种为同属的偏顶蛤,系统进化树中,远东偏顶蛤与同属的其他两个种聚为一支。本文研究结果为该种的遗传进化及多样性研究提供了基础数据。
关键词:远东偏顶蛤;ITS2;系统进化
远东偏顶蛤Modiolus kurilensis F.R.Bernard,1983 是广泛分布于中日韩俄海域的大型双壳贝类,隶属于瓣鳃纲、贻贝目、贻贝科、偏顶蛤属,由于其形态学特征与来自欧洲的偏顶蛤 Modiolus modiolus (Linnaeus,1758) 模式标本非常近似,致使很多人误将远东偏顶蛤当成偏顶蛤。2014年作者通过线粒体COI基因分析,确认大连黄海北部所得标本为远东偏顶蛤[1]。
真核生物核糖体 DNA (rDNA) 在染色体上的排列顺序为 18S、5.8S、28S rDNA,其中18S与5.8S rDNA之间存在着一个转录间隔区被称为第一转录间隔区(ITS1),而5.8S与28S rDNA之间的间隔区被称为第二转录间隔区(ITS2)。两个转录间隔区与18S 、5.8S 和 28S rDNA 一起在基因组中组成多拷贝转录单元。其中两个转录间隔区由于不加入成熟核糖体,进化过程中的选择压力小,进化速度较快,因而用于多种生物的进化关系构建、遗传变异、近似种类的区分及杂交种的鉴定[2-5]。本文首次测定并报道了远东偏顶蛤大连群体的ITS2 序列全长,并与近缘物种进行了初步比较分析,旨在为该种的系统发育、遗传多样性及遗传结构研究提供基础数据。
1材料与方法
1.1实验材料
本文所用实验材料于2015年8月采自辽宁省大连市付家庄海域。个体解剖后,取闭壳肌保存于95%乙醇溶液中。
1.2试验方法
剪取适量样本,去离子水浸泡以去除乙醇,采用TIANamp Marine animals DNA kit进行基因组DNA提取。
两个转录间隔区的PCR扩增引物序列分别为ITS-2A/ITS-2B:5' –GGGTCGATGAAGAA CGCAG -3' / 5' -GCTCTTCCCGCTTCACTCG-3'[6]。PCR扩增体系为:TaKaRa LA Taq(5 U/μL)0.5 μL,10×LA Taq Buffer II(Mg2+ Plus)5 μL,dNTP Mixture(2.5 mM each)8 μL,DNA模板50 ng,正反引物终浓度均为1.0 μM。PCR扩增程序为:94 ℃预变性 2 min;33 个循环包括94 ℃/30 s,52 ℃/30 s,72 ℃/1 min;最终72 ℃/5 min 延伸。扩增所得片段经琼脂糖电泳检测后送交上海生工行克隆测序。
从GenBank中下载贻贝科中其他物种的ITS2序列,去掉两端5.8S及28S序列,用于比较分析,用DNAMAN计算远东偏顶蛤与其他物种ITS2序列的一致性,以虾夷扇贝(Mizuhopecten yessoensis)的ITS2序列为外群,采用PHYLIP软件以邻位相连法(neighbor joining) 进行系统发育树(bootstrap=1 000)。
2结果与讨论
2.1远东偏顶蛤ITS2序列
本实验共进行了四个个体的ITS2 PCR扩增,扩增得到的片段约500 bp(图1),经克隆测序,去除片段两端的5.8S及28S序列,得到长度为245 bp的ITS2全序列,四个个体扩增片段序列完全一致,碱基序列及长度无多态性(图2)。
2.2远东偏顶蛤与其他物种ITS2序列的比较
利用DNAMAN计算了远东偏顶蛤与贻贝科其他物种及虾夷扇贝ITS2序列的一致性,结果列于表1。结果显示:贻贝科内物种ITS2长度在242-386bp之间,与远东偏顶蛤一致性最高的物种为其同属的的两个物种,其中Modiolus modiolus与远东偏顶蛤相比仅有3个碱基的缺失和3个碱基的突变。虽然贻贝Mytilus_edulis与远东偏顶蛤同属一科,然而其ITS2一致性小于非同科的虾夷扇贝,可能是由于贻贝的ITS2序列过长,导致了其与远东偏顶蛤序列一致性的严重降低。
ITS2在进化过程中选择压力较小,致使其长度及序列差异不仅存在于种间,还存在于同种的不同种群、同群体的不同个体间,甚至同一个体也存在碱基的突变[7]。喻达辉等在研究珠母贝属 6个种的 ITS 2分子标记时曾发现同种不同个体间存在少数碱基突变现象[8]。本研究虽然尚未发现个体间ITS2碱基的差异,但由于本实验仅以一个群体的四个样本为材料,不能武断地认为该种ITS序列中不存在差异。
2.3贻贝科ITS2系统进化树的构建
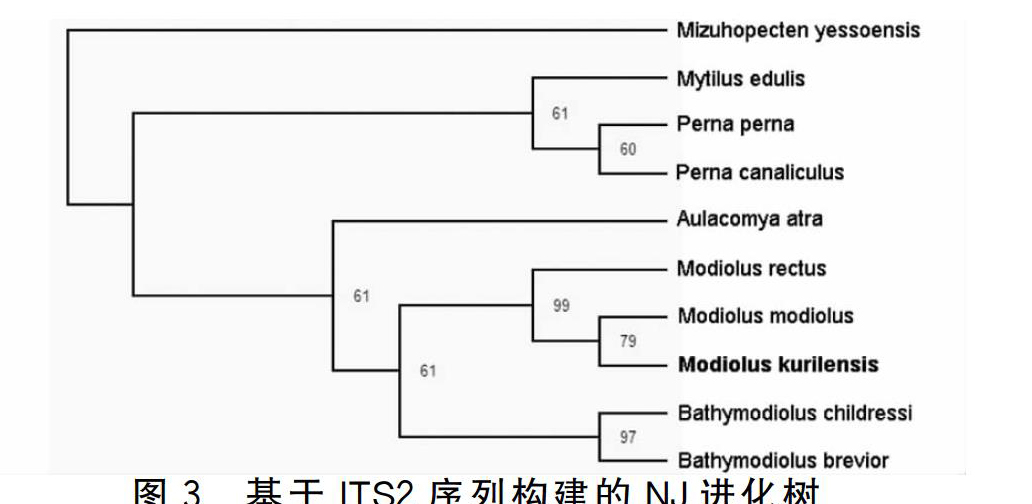
在利用上述ITS2序列构建的系统进化树中,同属的物种基本分布于同一姊妹分支上,东方偏顶蛤与同属的Modiolus modiolus 和Modiolus rectus聚为一支,而贻贝与同一亚科的Perna属的两个物种也分布在较近的分支上(图3)。虽然个别分支置信度仅为60%,但其显示出的进化关系与传统的形态学分类结果保持一致。上述系统进化树显示了该科进化的总体趋势:贻贝科进化分大分支,以贻贝亚科为代表的为一大分支,而另一分支则以偏顶蛤亚科代表。这一结果与Distel[9]使用18S序列研究该科进化的结果相似。
图3基于ITS2序列构建的NJ进化树
作者曾经利用东方偏顶蛤ITS1序列进行进化树构建,发现无论长度还是碱基顺序,不同物种ITS1均显示出很大的差异,以此构建的进化树与已有的分类系统大相径庭,差异很大,而本文用ITS2序列构建的系统进化树则与现有分类系统吻合的较好。孟学平等[10]曾经使用ITS2序列构建了蛤蜊科 3 种贝类的系统进化树,其显示的进化关系与使用16S序列构建的相似。综上所述,与ITS1相比,用ITS2构建的进化树能更好地反映近缘物种的进化关系。
参考文献:
[1] LIU Weidong,HE Jing,BAO Xiangbo.Mitochondrial DNA Supports the Validity of Modiolus kurilensis F.R.Bernard,1983.Shell Discoveries [J],2014 (3): 18-19
[2] Weekers PHH,De Jonckheere FJ,Dumont HJ.Phylogentic relationships inferred from ribosomal ITS sequences and biogeographics patterns in representative of the genus Calopteryx (Insecta: Odonata) of the West Mediterranean and adjacent west European zone [J].Mol Phyl Evol,2001,20: 89-99
[3] Chilton NB,Newton LA,Beveridge I,Gasser RB.Evolutionary relationships of trichostrongyloid Nematodes (Strongylida) inferred from ribosomal DNA sequence data [J].Mol Phyl Evol,2001,19: 367-386
[4] 孙桂金,潘杰,刘可春,等.2011.毛蚶、泥蚶和魁蚶 ITS1 核苷酸序列分析[J].生物技术通报,2011(7):117-120
[5] 龚理,徐晖,李军,等.褐牙鲆(♀)、夏鲆(♂)及其杂交子一代的 ITS1 序列特征分析[J].中国水产科学,2015,22(1): 17-23
[6] Hedgecock D,Li G ,Banks M A,et al.Occurrence of the Kumamoto oyster Crassostrea sikamea in the Ariake Sea,Japan [J] .Marine Biology.1999,133(1):65-68.
[7] 唐伯平,周开亚,宋大祥.核rDNA ITS 区序列在无脊椎动物分子系统学研究中的应用[J].动物学杂志,2002,37(4):67-72
[8] 喻达辉,朱嘉濠.珠母贝属6个种的 ITS 2分子标记研究[J].南方水产,2005,1(4):6-12.
[9] Distel DL.Phylogenetic Relationships among Mytilidae (Bivalvia): 18S Rrna Data Suggest Convergence in Mytilid Body Plans [J].Molecular Phylogenetics and Evolution.2000,15(1):25–33
[10] 孟学平,高如承,董志国,等.蛤蜊科 3 种贝类 16S rRNA 基因片段及 ITS2 核苷酸序列分析[J].湛江海洋大学学报,2006,26(4):8-13

