核酸外切酶辅助的信号放大策略在生化分析中的应用进展
文立 徐凤州 何晓晓 王柯敏何定庚 卿太平 邹振

摘 要 作为工具酶重要成员之一的核酸外切酶是一类无严格碱基序列依赖性的水解酶。近年来,通过利用核酸外切酶水解方式的差异性与纳米技术、酶切循环效应、核酸适配体技术、金属离子稳定的非沃森-克里克碱基配对系统、核酸荧光染料探针技术、电化学方法等相结合,发展了一系列核酸外切酶辅助的信号放大技术,对于提高生化分析检测方法与技术的灵敏度起到了非常关键的作用,已广泛应用于核酸、蛋白质、离子和小分子等物质的高灵敏检测。为了更好地理解和应用核酸外切酶辅助的信号放大策略,本文对核酸外切酶发展的信号放大策略在生化分析中的应用进展进行了综述。
关键词 分析检测;工具酶;信号放大;核酸外切酶;评述
1 引 言
快速、准确、灵敏地检测生化样品中的离子、分子、核酸、蛋白等对疾病的预防诊断、环境监测、食品监控等具有十分重要的意义。通常,实际生化分析样品中仅存在痕量靶物质,因此高灵敏度和高准确性一直是分析检测方法的改进目标。针对这一需求,除了直接发展高灵敏的方法外,放大检测法能降低分析方法或传感器的设计难度以及对专业大型仪器的依赖。根据放大的对象,放大检测法可以简单分为三类:靶标放大策略、探针放大策略和信号放大策略[1]。靶标放大策略通常是通过一定的手段特异性复制靶标,使其达到常规方法可以检测的水平。如经典的靶标放大策略——聚合酶链式反应(Polymerase chain reaction, PCR)可以实现108~109倍的靶标放大[2];探针放大策略,靶标的量不变,探针序列被不断放大,如滚环放大技术(Rolling circle amplification, RCA),它可以实现103~104倍左右的信号增强[3]。
相对于靶标放大策略和探针放大策略,信号放大策略有更多的可选性,如近年来发展的通过杂交链式反应、工具酶、酶联催化反应、纳米材料等生物及化学方法放大分析与传感器界面的响应信号,可特异性地提高检测信号,降低背景及噪音信号,对于提高生化分析检测方法与技术的灵敏度起到了非常关键的作用[4~9]。在这些信号放大策略中,基于工具酶(如聚合酶、连接酶、限制性内切酶、切割内切酶、核酸外切酶、核糖核酸酶等)辅助的信号放大技术,由于具有操作简便、灵敏度和特异性高、反应条件温和、反应时间相对较短等优点,近年来在生化分析方面的得到了迅速发展。尤其是利用核酸酶水解方式的差异性与纳米技术、酶切循环效应、核酸适配体技术、金属离子稳定的非沃森-克里克碱基配对系统、核酸荧光染料探针技术、电化学方法等相结合发展的一系列核酸酶辅助的信号放大技术,已广泛应用于核酸、蛋白质、离子和小分子等物质的检测[10~14]。基于核酸酶辅助的信号放大策略主要有两大类,一是序列依赖性的限制性内切酶或切割内切酶辅助的信号放大策略,另一类是无序列依赖性的核酸外切辅助的信号放大策略。从核酸酶辅助的信号放大策略的通用性来讲,没有序列依赖性的核酸外切酶在探针设计和靶标分析方面更加简便易行,因而显示了更广的应用前景。本文即在简单概括核酸外切酶的分类以及基本特性的基础上,着重介绍核酸外切酶辅助的信号放大策略在核酸、蛋白质、小分子及离子分析检测中的研究进展,并展望其发展趋势。
2 核酸外切酶的分类及其基本特性
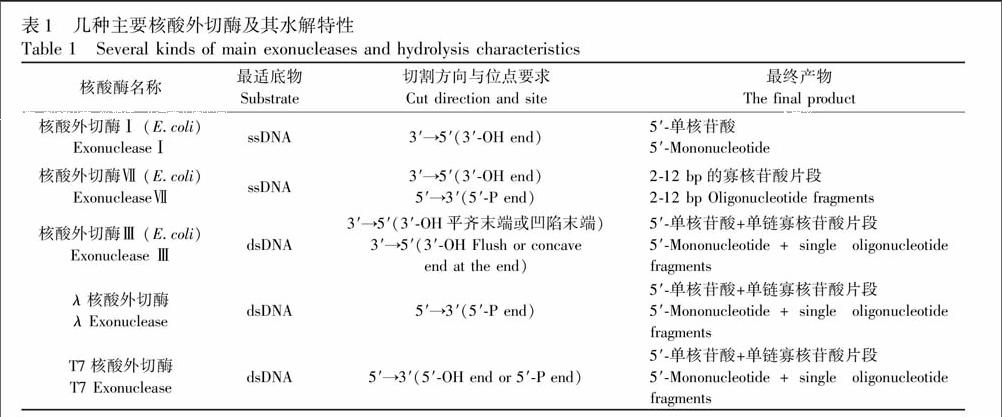
核酸外切酶简称外切酶(Exonuclease),是一类从多核苷酸链的一端开始按序催化水解3,5-磷酸二酯键,获得单个的核苷酸为最终产物(DNA为dNMP,RNA为NMP)的酶。根据其作用底物的特异性可将核酸外切酶分为单链的核酸外切酶和双链的核酸外切酶,如大肠杆菌核酸外切酶Ⅰ(Exo Ⅰ)和核酸外切酶Ⅶ(Exo Ⅶ)属于单链的核酸外切酶,而双链的核酸外切酶则包括了大肠杆菌核酸外切酶Ⅲ(Exo Ⅲ)、λ噬菌体核酸外切酶(λ exo)以及T7核酸外切酶(T7 exo)等。尽管不像核酸内切酶那样具有对催化水解底物存在较强的碱基序列依赖性,不同的核酸外切酶存在不同的水解条件,可以区别出从分子链的3′末端或5′末端开始切断和对单链DNA或双链DNA具有特异性作用。为了更好地理解和应用核酸外切酶辅助的信号放大策略,本文总结了在生物分析研究中应用较多的几种核酸外切酶,如表1和图1所示。以核酸外切酶Ⅲ为例,该酶的切割底物为双链DNA,切割方向为3′→5′,且要求3′端为平齐或者凹陷末端,切割后产物为5′-单核苷酸。
3 核酸外切酶辅助的信号放大策略在生化分析中的应用
3.1 核酸检测
核酸是组成生命体的最主要生物大分子之一,是细胞内携带遗传信息的物质,它控制着蛋白质的合成和有机体细胞的机能,在生物的生长、发育、繁殖、遗传和变异等生命活动中占有极其重要的地位[16~18]。核酸序列中如果出现一个或几个核苷酸的取代、缺失或插入等微小的改变,都将引起基因突变或多态性,导致遗传病或其他疾病的出现[19,20]。因此,对人体的血液、组织及体液等样品中特定基因序列和与遗传疾病相关的单碱基突变的检测分析,在分子生物学、疾病早期诊断与治疗、发病机理研究以及药物筛选等方面都具有重大意义[21~25]。
最早的核酸检测方法是基于核酸杂交原理的分析方法,主要包括有Southern印迹杂交、Northern印迹杂交和原位杂交等[26~28]。然而当靶标序列浓度很低时,这种直接杂交技术的检测效果比较差,灵敏度低,大大限制了其广泛应用。随着生物技术的不断发展,一些新型的放大检测技术不断涌现。如体外扩增技术[29~31]、酶联催化反应[32,33]、脱氧核酶催化[34,35]、核酸酶辅助等[36~39]。其中,核酸外切酶辅助的靶标循环技术应用尤为广泛[40~44]。
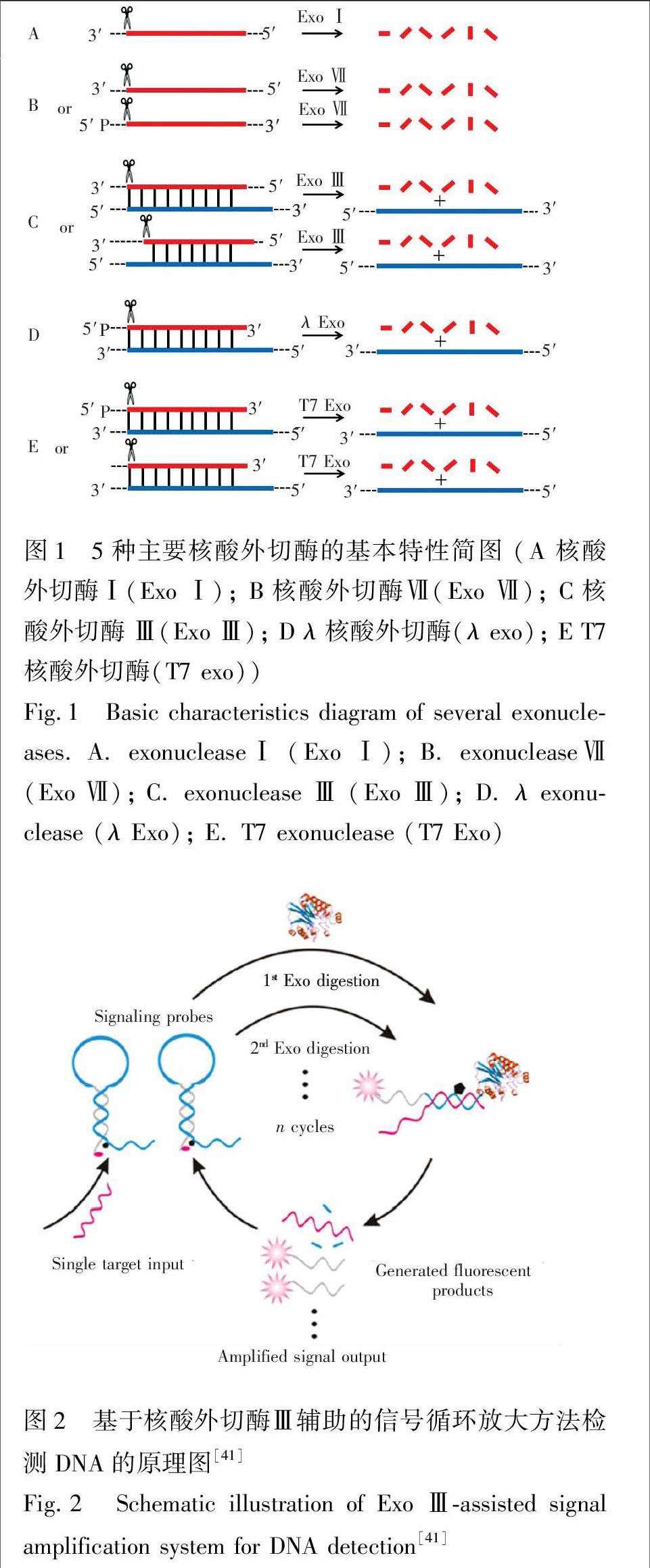
2010年,Zuo等[41]使用分子信标作为信号探针,发展了一种核酸外切酶Ⅲ辅助的靶标循环信号放大技术用于DNA的高灵敏检测。实验原理如图2所示,在分子信标的5′端标记荧光团,与5′端平齐的3′端中间位置标记淬灭基团(分子信标3′端有7个碱基的突出序列用于抵抗核酸外切酶Ⅲ的水解),形成发夹后,荧光基团和淬灭基团距离非常近,从而使荧光基团的荧光被淬灭。当目标DNA存在时,分子信标被打开形成能被核酸外切酶Ⅲ水解的3′平末端,核酸外切酶Ⅲ沿双链的3′→5′方向逐步切除单核苷酸,并释放出荧光基团。而同时释放的目标DNA能与另一个分子信标杂交,引起酶切循环信号放大,这种方法对DNA的检出限达到20 amol/L。
相比昂贵的核酸探针标记,免标记的方法更简便、廉价。基于类似的原理,Xu等[42]利用λ核酸外切酶辅助的信号放大作用设计了一种超灵敏的电化学传感器。通过铁氰根离子在靶标存在与否情况下与电极的远近,表现出不同的电化学阻抗来特异性检测乳腺癌基因BRCA1。此外,Kong等[43]将核酸外切酶辅助的靶标循环与金纳米颗粒联用,实现双重放大免标记检测DNA,该方法检出限可以达到0.6 pmol/L。
除了对样品中DNA的检测外,RNA尤其是microRNA (miRNA)(被视为一种新型的肿瘤标志物)的检测也非常重要。Wang等[44]利用电化学方法,结合T7核酸外切酶的循环放大作用,设计了一种超灵敏检测miRNA的方法。其原理如图3所示:首先将能与miRNA互补的DNA探针的3′端修饰于金电极表面,此时由于表面覆盖有大量DNA使得阻抗信号较大。当加入miRNA之后,miRNA与DNA探针形成双链,在T7核酸外切酶的作用下,修饰在金电极上的DNA探针从5′端开始被水解,同时释放出的靶标miRNA能与下一个捕获探针杂交,实现循环放大。最后,金电极表面的DNA探针被剪切掉,导致电化学阻抗信号降低,进而实现miRNA的高灵敏检测。
图3 基于T7核酸外切酶辅助的信号循环放大方法检测miRNA的原理图[44]
Fig.3 Schematic illustration of T7 exo-assisted signal amplification system for miRNA detection[44]
此外,核酸序列中如果出现一个或几个核苷酸的改变,都将引起基因突变,导致遗传或其它疾病的出现。其中单核苷酸多态性(Single nucleotide polymorphisms, SNPs)占所有多态性的90%以上,是频率最高、信息含量最多的一种变异,也是最多见的遗传变异类型[45,46], 如镰刀形红细胞贫血、β-地中海贫血、囊性纤维化病以及肿瘤等[47,48]。由于单核苷酸多态性通常只是核酸序列中一个核苷酸改变,类似非靶标核酸的存在,因此针对核酸检测的一些方法也被用于与遗传疾病相关度较高的单碱基突变的检测分析。2014年,Wu等[49]利用分别标有荧光基团和淬灭基团分子探针来特异性检测单核苷酸多态性。最近,Wu等[50]为了避免分子探针双标效率低,费用昂贵等缺点,结合金颗粒和核酸外切酶Ⅲ设计了一种比色方法, 用于区分单碱基错配。
3.2 蛋白质检测
蛋白质是基因表达的最终产物,是细胞内各类代谢和调控等生命功能的执行者,也是致病因子、药物对机体作用的最重要的靶标分子。人类的绝大多数疾病都是由蛋白质表达异常引起的,蛋白的表达指示了细胞的生理和病理状态,揭示了药物及环境因素对生命体的影响。因此,系统性的蛋白质研究和分析对理解生命现象、监测疾病进程和药物作用等方面都具有非常重要的意义[51,52]。
基于传统的免疫分析是常规的蛋白质检测方法,通常在抗原或抗体上标记放射性物质、酶或荧光素等,然后利用抗原-抗体间的特异性反应来实现对蛋白质的灵敏特异性检测[53~59]。虽然免疫分析可以对蛋白质实现高灵敏度检测,但以上技术实验过程复杂,操作繁琐,效率低,不适合系统化、大规模的蛋白质组学研究,且功能化修饰可能影响抗原抗体间的亲和力。因此,发展一些简单、快速、灵敏的蛋白质分析新方法显得尤为重要。
近年来,基于蛋白与核酸的作用以及核酸与工具酶的作用,发展了一系列新型的蛋白质分析方法[60~64]。这些方法不仅避免了传统蛋白质检测方法中操作复杂,常需对蛋白质进行标记,不易保持其生物活性等缺点,还有效地提高了蛋白质检测的效率与灵敏度。2010年,Ou等[60]基于TATA结合蛋白(TBP)与含有TATA序列的DNA的特异结合能有效抵抗核酸外切酶的作用,设计了一种核酸外切酶Ⅲ辅助的信号放大比色检测蛋白质的新方法。如图4所示,首先在不同的金纳米颗粒上分别修饰上能互补的单链DNA,当两种金纳米颗粒按一定比例混合后,互补DNA之间的碱基作用力使得金纳米颗粒相互靠近,从而产生金纳米颗粒团聚效应,肉眼看到金纳米颗粒溶液由酒红色变成暗紫色。然后,加入核酸外切酶Ⅲ,当TBP存在时,因TBP与含有TATA序列DNA特异性结合,能对DNA 3′端有保护作用(空间位阻效应),使得核酸外切酶Ⅲ不能切割DNA链,从而金颗粒溶液一直呈暗紫色;当TBP不存在时,DNA链失去蛋白的保护作用,核酸外切酶Ⅲ从DNA 3′端切割,使得金颗粒彼此远离,溶液呈酒红色,从而实现对DNA特异结合蛋白TBP的比色检测。
图4 基于核酸外切酶Ⅲ辅助的信号循环放大方法和金颗粒检测DNA特异结合蛋白的原理图[60]
Fig.4 Schematic illustration of Exo Ⅲ-assisted signal amplification system and gold nanoparticles for sequence-specific DNA-binding protein detection[60]
随着更多的靶标蛋白的核酸适配体成功筛选,基于适配体作为识别元件的核酸外切酶辅助信号放大的蛋白质检测也备受关注。Chen等[61]发明了一种基于核酸适配体和核酸外切酶Ⅲ辅助下循环放大的平台用于检测溶菌酶。实验原理如图5所示,设计一个发夹结构DNA,DNA链的一部分为溶菌酶的核酸适配体。当有溶菌酶存在的情况下,探针发生空间构象变化使得非核酸适配体部分暴露出,然后加入标记了荧光基团的信号探针,该探针与非核酸适配体部分互补配对,形成平齐末端的DNA双链,在核酸外切酶Ⅲ的作用下,信号探针被剪切,释放出荧光基团,被打开的发夹探针可以循环利用,释放出大量荧光基团,由于荧光分子无法被吸附到氧化石墨烯表面,从而呈现出强的荧光信号。当不存在溶菌酶时,发夹结构无法打开,信号探针无法与其互补结合,核酸外切酶Ⅲ也无法切割,荧光基团一直被标记在信号探针上,信号探针与氧化石墨烯结合,荧光基团被氧化石墨烯淬灭,呈现很低的荧光信号。
基于类似的原理,凝血酶与其对应的核酸适配体结合后构型会发生变化,Wang等[62]设计了一种检测凝血酶的方法,该方法特异性好,灵敏度高,可达到对几个蛋白分子的准确定量。此外,利用一些核酸外切酶对底物的偏好性(如磷酸化、甲基化等),发展了一些磷酸激酶、磷酸化酶、甲基转移酶等的检测新方法。Liu等[63]利用λ核酸外切酶对底物5′磷酸化的要求,结合氧化石墨烯平台设计了一种快速检测T4多核苷酸激酶活性的方法。
图5 基于氧化石墨烯和核酸外切酶Ⅲ辅助的信号循环放大方法检测溶菌酶的原理图[61]
Fig.5 Schematic illustration of GO and Exo Ⅲ-assisted signal amplification system for lysozyme detection[61]
3.3 生物小分子检测
除了核酸、蛋白质等生物大分子外,一些生物活性小分子在机体的新陈代谢及生命过程中也起着重要作用,与人类的健康息息相关。如三磷酸腺苷(ATP)发生水解时,能产生大量供给人体的日常所需的生命活动的能量;还原型的谷胱甘肽(GSH)可以通过与过多的自由基或者对身体有害处的重金属等物质结合,以达到中和有毒物质的作用,并能够将之排出体外,所以GSH是人体内部特别的抗氧化剂跟自由基清除剂。以GSH、ATP等为代表的细胞内小分子活性物质的检测对研究机体的生理功能和疾病早期诊断具有重要意义。目前,用于生物活性分子的分析检测手段主要有荧光、比色、电化学以及同位素标记等。其中,利用荧光方法分析小分子具有灵敏度高、分析快捷、样品制备简单等优点而被广泛关注。随着配体指数富集系统进化技术(SELEX)的发展,一些小分子的核酸适配体(Aptamer)被发现,利用其适配体与小分子的特异性结合,已发展了许多新的小分子荧光分析新方法[65~67]。工具酶对作用底物的结构有较强的依赖性,而这些小分子与核酸适配体的结合又能诱导核酸适配体构型的变化,因而,一些工具酶也被广泛用于小分子的放大检测[68~71]。
如图6所示,Xu等[68]设计了一种基于核酸外切酶Ⅲ催化的目标物循环放大方法免标记检测ATP。利用ATP的核酸适配体构建了一个发夹探针,且使得茎部3′端突出,以避免被核酸外切酶Ⅲ降解。当有ATP存在时,ATP与发夹探针中的核酸适配体序列结合,使得核酸适配体的构型发生变化,导致茎部3′端凹陷,加入核酸外切酶Ⅲ后即可从3′端开始剪切双链部分,最终形成单链DNA,并释放出ATP,而ATP即可继续循环利用,实现循环放大,双链特异性染料无法嵌入循环产生的单链DNA,因此没有荧光信号。而没有目标ATP存在时,发夹探针的茎部双链部分能嵌入SYBR Green I呈现出非常高的荧光信号。
Zheng等[69]利用核酸适配体与靶标结合产生的空间位阻作用达到抗酶切的效果,设计了一种基于核酸外切酶Ⅰ辅助的分析检测平台,可用于离子、小分子物质及蛋白的检测。
图6 基于核酸外切酶Ⅲ辅助的目标物循环放大方法检测ATP的原理图[68]
Fig.6 Schematic illustration of Exo Ⅲ-assisted target amplification system for ATP detection[68]
3.4 离子检测
重金属离子Hg2+和Ag2+由于其性质相对比较稳定,在环境中很难被生物降解,且生物毒性高,因此,重金属污染严重危害环境及生物的生存。由于重金属污染以及重金属随着食物链的富集作用,使得重金属离子广泛存在于水、土壤和食物当中,
图7 基于核酸外切酶Ⅲ辅助的信号放大方法检测Hg2+的原理图[80]
Fig.7 Schematic illustration of Exo Ⅲ-assisted signal amplification system for Hg2+ detection[80]
然而重金属的摄入会直接损害人体健康,过量甚至会危及生命[72,73],因此针对重金属离子的监测显得极为重要。传统的检测方法是基于专业仪器的原子吸收光谱、原子发射光谱以及电感耦合等离子体质谱法等,这些方法存在操作复杂、费时费力、成本高等缺点。近些年,一些基于小分子发色团、纳米材料、核酶以及聚合物材料的重金属离子传感检测方法被不断发展[74~76]。同时,基于DNA的特殊碱基或位点能特异性地与某些金属离子结合,形成非自然碱基对(即金属离子稳定的非沃森-克里克碱基配对系统,如“T-Hg2+-T”、“C-Ag+-C”),许多针对相应离子的传感器被发展出来[77~79]。正是由于这些离子与核酸的结合,许多工具酶,特别是核酸外切酶也被引入相应离子的放大检测体系[80,81]。如Chen等[80]发明了一种基于核酸外切酶Ⅲ辅助的信号放大方法可视化检测Hg2+的一次性条带生物传感器,其原理如图7所示,发夹DNA和辅助DNA中间部分可互补配对,其中发夹DNA的3′端和辅助DNA的5′均为富T序列,没有Hg2+存在时,二者无法杂交以打开发夹DNA;只有当Hg2+存在时,通过T-Hg2+-T非自然配对使得形成具有平齐末端的双链DNA,进而被核酸外切酶Ⅲ识别并从3′端开始剪切,释放出的辅助DNA和Hg2+继续循环利用,产生大量ssDNA。在B部分中,结合部分(Conjugate pad)含有大量修饰了DNA的金颗粒(AuNPs-DNA probe1),检测条带区域(TZ)处含有修饰了链霉亲和素和生物素的DNA(SA-biotin-DNA probe2),对照条带区域(CZ)处含有修饰了链霉亲和素和生物素的另一条DNA(SA-biotin-DNA probe3)。其中AuNPs-DNA probe1与SA-biotin-DNA probe3是互补的,只要条带部分完好,金颗粒被富集于CZ条带处显色,该条带可以检验条带传感器是否完好。当检测体系中有Hg2+存在时,产生的ssDNA可以与AuNPs-DNA probe1和SA-biotin-DNA probe2杂交,从而使金颗粒富集于TZ条带处显色;而当没有Hg2+存在时,无法产生ssDNA,因此TZ条带处不能显色。该方法方便快捷,可直接肉眼观察到结果,且灵敏度高,检出限可达1 pmol/L。类似T-Hg2+-T非自然碱基配对,基于C-Ag+-C效应和核酸外切酶Ⅲ辅助放大,Xu等[79]设计了一种的循环放大方法检测Ag+,其检出限可达0.03 nmol/L。
4 展 望
核酸外切酶在生化分析领域的应用发展极其快速而且应用方式变化层出不穷。纵观核酸外切酶在生化分析中的应用情况,其在生化分析中的应用发展未来将有可能呈现出以下趋势,包括:(1)多元化信号获取方式。可以通过改进仪器达到高灵敏的检测(如荧光信号、电化学信号等),也可通过肉眼直接观察的比色方法以及试纸条显色等方法得出结果,未来可能有更多、更灵敏的信号输出方式;(2)酶切循环放大和功能化纳米材料信号放大相结合实现多重放大。例如酶和金属纳米颗粒结合应用,先通过酶切实现信号放大,然后通过结合金属纳米颗粒二级放大实现更灵敏的检测; (3)向生化传感的实用性方向发展。利用核酸外切酶来实现信号放大的传感器通常都比较轻巧,不需要配备严格的实验条件和贵重的仪器,可用于发展便携的传感器或检测试纸条等,因此这类传感器有望实现即时检测(POCT)和临床应用;(4)复杂生物样品的检测。基于核酸外切酶的放大检测方法,目前还主要应用于体外缓冲液样品的检测,随着更多的离子、分子、细胞等与核酸结合的发现,核酸外切酶也将用于更多样性的分析物的检测,其难点和挑战是在更为复杂的生物样品(如体液、细胞等)中实现目标物的检测。我们相信,随着对核酸外切酶性质和应用的进一步探索,核酸外切酶在生化分析领域将会有更加广阔的应用前景。
References
1 Gerasimova Y V, Kolpashchikov D M. Chem. Soc. Rev., 2014, 43(17): 6405-6438
2 Minunni M, Tombelli S, Fonti J, Spiriti M M, Mascini M, Bogani P, Buiatti M. J. Am. Chem. Soc., 2005, 127(22): 7966-7967
3 Banér J, Nilsson M, Mendel-Hartvig M, Landegren U. Nucleic Acids Res., 1998, 26(22): 5073-5078
4 Tao C, Yan Y, Xiang H, Zhu D, Cheng W, Ju H, Ding S. Chem. Commun., 2015, 51(20): 4220-4222
5 Wu Z K, Zhou D M, Wu Z, Chu X, Yu R Q, Jiang J H.Chem. Commun., 2015, 51(14): 2954-2956
6 Sun J, Jiang W, Zhu J, Li W, Wang L. Biosens. Bioelectron., 2015, 70: 15-20
7 Bao T, Wen W, Zhang X, Xia Q, Wang S. Biosens. Bioelectron., 2015, 70: 318-323
8 Liu S, Liu T, Wang L.Chem. Commun., 2015, 51(1): 176-179
9 Liu X, Li W, Hou T, Dong S, Yu G, Li F. Anal. Chem., 2015, 87(7): 4030-4036
10 Guo X, Liu P, Yang X, Wang K, Wang Q, Guo Q, Huang J, Liu J, Song C, Li W. Analyst, 2015, 140(6): 2016-2022
11 Guo X, Yang X, Liu P, Wang K, Wang Q, Guo Q, Huang J, Li W, Xu F, Song C. Analyst, 2015, 140(6): 4291-4297
12 Liu P, Sun S, Guo X, Yang X, Huang J, Wang K, Wang Q, Liu J, He L. Anal. Chem., 2015, 87(5): 2665-2671
13 Miyake Y, Togashi H, Tashiro M, Yamaguchi H, Oda S, Kudo M Y, Kondo Y, Sawa R, Fujimoto T, Machinami T, Ono A. J. Am. Chem. Soc., 2006, 128(7): 2172-2173
14 Liu Y , Luo M, Xiang X, Chen C, Ji X, Chen L, He Z. Chem. Commun., 2014, 50(20): 2679-2681
15 GUO Qiu-Ping, ZHAO Xia-Yu, XIE Qin, WANG Ke-Min, WAN Jun, YUAN Bao-Yin, TAN Yu-Yu. Chem. J. Chinese Universities, 2014, 35(8): 1646-1651
郭秋平, 赵下雨, 谢 琴, 王柯敏, 万 俊, 袁宝银, 谭誉宇. 高等学校化学学报, 2014, 35(8): 1646-1651
16 Woychik R P, Klebig M L, Justice M J, Magnuson T R, Avrer E D. Mutat. Res-Fund Mol. M., 1998, 400(1): 3-14
17 Williams G T, Smith C A. J. Cell Sci., 1993, 74(5): 777-779
18 ZHONG Bi-Ling,SONG Yong-Sheng. Chin. J. Cancer., 2000, 19(5): 432-435
钟碧玲, 宋永生. 中国癌症杂志, 2000, 19(5): 432-435
19 ZHENG Qin, LIU Hua, ZENG Ting-Ting, YE Yuan-Xin,JIANG Neng-Gang,SU Jun. Chinese Journal of Evidence-based Medicine, 2013, 13(2): 170-175
郑 沁, 刘 华, 曾婷婷, 叶远馨, 蒋能刚, 粟 军. 中国循证医学杂志, 2013, 13(2): 170-175
20 ZHAO Hong-Shan, HUANG Shang-Zhi. Chinese J Med Genet., 2000, 17(1): 6-9
赵红珊, 黄尚志. 中华医学遗传学杂志, 2000, 17(1): 6-9
21 Osada M, Ohba M, Kawahara C, Ishioka C, Kanamaru R, Katoh I, Katoh I, kawa Y, Nimura Y, N akagawara A, Obinata M I, Kawa S. Nat. Med., 1998, 4(7): 839-843
22 Shi S T, Yang G Y, Wang L D, Xue Z, Feng B, Ding W, Xing E P, Yang C S. Carcinogenesis, 1999, 20(4): 591-597
23 CAI Wei-Ming, HU Min-Jun. Zhejiang Journal of Integrated Traditional Chinese and Western Medicine, 2002, 12(11): 661-666
蔡卫民, 胡敏君. 浙江中西医结合杂志, 2002, 12(11): 661-666
24 ZENG Yi-Tao. Chinese J Hematol., 1996, 17(8): 393-394
曾溢滔. 中华血液学杂志, 1996, 17(8): 393-394
25 Marton M J, DeRisi J L, Bennett H A, Iyer V R, Meyer M R, Roberts C J, Stoughton R, Burchard J, Slade D, Dai H Y, Bassett Jr D E, Hartwel L H, Brown, P O, Friend, S H. Nat. Med., 1998, 4(11): 1293-1301
26 Mulbry W W, Karns J S, Kearney P C, Nelson J O, McDaniel C S, Wild J. R. Appl Environ Microb., 1986, 51(5): 926-930
27 Correa R R, Mariash C N, Rosenberg M E. Biotechniques., 1992, 12(2): 154-158
28 Yang H, McLeese J, Weisbart M, Dionne J L, Lemaire I, Aubin R A.Nucleic Acids Res., 1993, 21(14): 3337-3338
29 Krokene P, Barnes I, Wingfield B D, Wingfield M J. Mycologia, 2004, 96(6): 1352-1354
30 Odin E, Larsson L, Aram M, Gustavsson B, Larsson P A. Tumor Biol., 1998, 19(3): 167-175
31 Pfaffl M W.Nucleic Acids Res., 2001, 29(9): e45-e45
32 De La Rica R, Stevens M M. Nat. Nanotechnol., 2012, 7(12): 821-824
33 Wang Q, Liu F, Yang X H, Wang K M, Liu P, Liu J B, Huang J, Wang H. Sensor Actuat B-Chem., 2013, 186: 515-520
34 Deng M, Zhang D, Zhou, Y, Zhou X. J. Am. Chem. Soc., 2008, 130(39): 13095-13102
35 Zhao X, Gong L, Zhang X B, Yang B, Fu T, Hu R, Tan W H ,Yu R Q. Anal. Chem., 2013, 85(7): 3614-3620
36 Yin B C, Liu Y Q, Ye B C.J. Am. Chem. Soc., 2012, 134(11): 5064-5067
37 Bi S, Zhang J, Zhang S.Chem. Commun., 2010, 46(30): 5509-5511
38 Li W, Wu P, Zhang H, Cai C. Anal. Chem., 2012, 84(17): 7583-7590
39 Ren W, Gao Z F, Li N B, Luo H Q. Biosens. Bioelectron., 2015, 63: 153-158
40 Guo Q, Yang X, Wang K, Tan W, Li W, Tang H, Li H. Nucleic Acids Res., 2009, gkn1024
41 Zuo X, Xia F, Xiao Y, Plaxco K W.J. Am. Chem. Soc., 2010, 132(6): 1816-1818
42 Song C, Yang X, Wang K, Wang Q, Liu J, Huang J, He L, Liu P, Qing Z H, Liu W. Chem. Commun., 2015, 51(10): 1815-1818
43 Kong R M, Song Z L, Meng H M, Zhang X B, Shen G L, Yu R Q. Biosens. Bioelectron., 2014, 54: 442-447
44 Wang M, Fu Z, Li B, Zhou Y, Yin H, Ai S. Anal. Chem., 2014, 86(12): 5606-5610
45 Risch N, Merikangas K. Science, 1996, 273(5281): 1516-1517
46 Kruglyak L. Nat Genet., 1997, 17(1): 21-24
47 WU Guan-Yun, WANG Shen-Wu. Gene Diagnosis. Beijing: People′s Medical Publishing House, 1988: 131-142
吴冠芸, 王申五. 基因诊断. 北京: 人民卫生出版社, 1988: 131-142
48 WU Guan-Yun, FANG Fu-De. Gene Diagnosis Technology and Application. Beijing: Beijing Medical University and China Xie-He. Medical University Joint Publishing House, 1992: c176-177
吴冠芸, 方福德. 基因诊断技术及应用, 北京: 北京医科大学、北京协和医科大学联合出版社, 1992: c176-177
49 Wu T, Xiao X, Zhang Z, Zhao M. Chem. Sci., 2015, 6(2): 1206-1211
50 Wu S, Liang P, Yu H, Xu X, Liu Y, Lou X, Xiao Y. Anal. Chem., 2014, 86(7): 3461-3467
51 Mikami T, Yanagisawa N, Baba H, Koike M, Okayasu I. Cancer, 1999, 85(2): 318-325
52 JING Li, ZHANG Jian-Zhong, GUO Feng-Ying. Chin. J. Cancer., 2003, 13(1): 27-29
景 丽, 张建中, 郭凤英. 中国癌症杂志, 2003, 13(1): 27-29
53 Ellington A D, Szostak J W. Nature, 1990, 346(6287): 818-822
54 Tuerk C, Gold L. Science, 1990, 249(4968): 505-510
55 Guo W M, Kong K W, Brown C J, Quah S T, Yeo H L, Hoon S, Seow Y.Mol. Ther-Nucl. acids., 2014, 3(12): e217
56 Famulok M, Hartig J S, Mayer G. Chem. Rev., 2007, 107(9): 3715-3743
57 Voller A, Bartlett A, Bidwell D E. J. Clin. Pathol., 1978, 31(6): 507-520
58 Blake M S, Johnston K H, Russell-Jones G J, Gotschlich E C. Anal. Biochem., 1984, 136(1): 175-179
59 Schaeffer M, Orsi E V, Wide lock D. Bact. Rev., 1964, 28(4): 402-408
60 Ou L J, Jin P Y, Chu X, Jiang J H, Yu R Q. Anal. Chem., 2010, 82(14): 6015-6024
61 Chen C, Zhao J, Jiang J, Yu R. Talanta, 2012, 101: 357-361
62 Wang X L, Li F, Su Y H, Sun X, Li X B, Schluesener H J, Tang F, Xu S Q. Anal. Chem., 2004, 76(19): 5605-5610
63 Liu L, Lei J, Gao F, Ju H X. Talanta, 2013, 115: 819-822
64 Li W, Liu X, Hou T, Li H, Li F., Biosens. Bioelectron., 2015, 70: 304-309
65 Kim Y S, Kim J H, Kim I A, Lee S J, Jurng J, Gu M B. Biosens. Bioelectron., 2010, 26(4): 1644-1649
66 HyunáKim T. Lab Chip, 2011, 11(1): 52-56
67 Deng C, Chen J, Nie L, Nie Z, Yao S. Anal. Chem., 2009, 81(24): 9972-9978
68 Xu Y, Xu J, Xiang Y, Yuan R, Chai Y. Biosens. Bioelectron., 2014, 51: 293-296
69 Zheng D, Zou R, Lou X. Anal. Chem., 2012, 84(8): 3554-3560
70 Wei Y, Chen Y, Li H, Shuang S, Dong C, Wang G. Biosens. Bioelectron., 2015, 63: 311-316
71 Sun J, Jiang W, Zhu J, Li W, Wang L. Biosens. Bioelectron., 2015, 70: 15-20
72 Renzoni A, Zino F, Franchi E. Environ. Res., 1998, 77(2): 68-72
73 Nichols J W, Brown S, Wood C M, Walsh P J, Playle R C. Aquat. Toxicol., 2006, 78(3): 253-261
74 Guo X, Qian X, Jia L. J. Am. Chem. Soc., 2004, 126(8): 2272-2273
75 Xue X, Wang F, Liu X. J. Am. Chem. Soc., 2008, 130(11): 3244-3245
76 Ono A, Togashi H. Angew. Chem. Int. Ed., 2004, 43(33): 4300-4302
77 Yin J, He X, Jia X, Wang K, Xu F. Analyst, 2013, 138(8): 2350-2356
78 Zhao C, Qu K, Song Y, Xu C, Ren J, Qu X. Chem-Eur J., 2010, 16(27): 8147-8154
79 Xu G, Wang G, He X, Zhu Y, Chen L, Zhang X. Analyst, 2013, 138(22): 6900-6906
80 Chen J, Zhou S, Wen J. Anal. Chem., 2014, 86(6): 3108-3114
81 Xuan F, Luo X, Hsing I M. Anal. Chem., 2013, 85(9): 4586-4593



摘 要 作为工具酶重要成员之一的核酸外切酶是一类无严格碱基序列依赖性的水解酶。近年来,通过利用核酸外切酶水解方式的差异性与纳米技术、酶切循环效应、核酸适配体技术、金属离子稳定的非沃森-克里克碱基配对系统、核酸荧光染料探针技术、电化学方法等相结合,发展了一系列核酸外切酶辅助的信号放大技术,对于提高生化分析检测方法与技术的灵敏度起到了非常关键的作用,已广泛应用于核酸、蛋白质、离子和小分子等物质的高灵敏检测。为了更好地理解和应用核酸外切酶辅助的信号放大策略,本文对核酸外切酶发展的信号放大策略在生化分析中的应用进展进行了综述。
关键词 分析检测;工具酶;信号放大;核酸外切酶;评述
1 引 言
快速、准确、灵敏地检测生化样品中的离子、分子、核酸、蛋白等对疾病的预防诊断、环境监测、食品监控等具有十分重要的意义。通常,实际生化分析样品中仅存在痕量靶物质,因此高灵敏度和高准确性一直是分析检测方法的改进目标。针对这一需求,除了直接发展高灵敏的方法外,放大检测法能降低分析方法或传感器的设计难度以及对专业大型仪器的依赖。根据放大的对象,放大检测法可以简单分为三类:靶标放大策略、探针放大策略和信号放大策略[1]。靶标放大策略通常是通过一定的手段特异性复制靶标,使其达到常规方法可以检测的水平。如经典的靶标放大策略——聚合酶链式反应(Polymerase chain reaction, PCR)可以实现108~109倍的靶标放大[2];探针放大策略,靶标的量不变,探针序列被不断放大,如滚环放大技术(Rolling circle amplification, RCA),它可以实现103~104倍左右的信号增强[3]。
相对于靶标放大策略和探针放大策略,信号放大策略有更多的可选性,如近年来发展的通过杂交链式反应、工具酶、酶联催化反应、纳米材料等生物及化学方法放大分析与传感器界面的响应信号,可特异性地提高检测信号,降低背景及噪音信号,对于提高生化分析检测方法与技术的灵敏度起到了非常关键的作用[4~9]。在这些信号放大策略中,基于工具酶(如聚合酶、连接酶、限制性内切酶、切割内切酶、核酸外切酶、核糖核酸酶等)辅助的信号放大技术,由于具有操作简便、灵敏度和特异性高、反应条件温和、反应时间相对较短等优点,近年来在生化分析方面的得到了迅速发展。尤其是利用核酸酶水解方式的差异性与纳米技术、酶切循环效应、核酸适配体技术、金属离子稳定的非沃森-克里克碱基配对系统、核酸荧光染料探针技术、电化学方法等相结合发展的一系列核酸酶辅助的信号放大技术,已广泛应用于核酸、蛋白质、离子和小分子等物质的检测[10~14]。基于核酸酶辅助的信号放大策略主要有两大类,一是序列依赖性的限制性内切酶或切割内切酶辅助的信号放大策略,另一类是无序列依赖性的核酸外切辅助的信号放大策略。从核酸酶辅助的信号放大策略的通用性来讲,没有序列依赖性的核酸外切酶在探针设计和靶标分析方面更加简便易行,因而显示了更广的应用前景。本文即在简单概括核酸外切酶的分类以及基本特性的基础上,着重介绍核酸外切酶辅助的信号放大策略在核酸、蛋白质、小分子及离子分析检测中的研究进展,并展望其发展趋势。
2 核酸外切酶的分类及其基本特性
核酸外切酶简称外切酶(Exonuclease),是一类从多核苷酸链的一端开始按序催化水解3,5-磷酸二酯键,获得单个的核苷酸为最终产物(DNA为dNMP,RNA为NMP)的酶。根据其作用底物的特异性可将核酸外切酶分为单链的核酸外切酶和双链的核酸外切酶,如大肠杆菌核酸外切酶Ⅰ(Exo Ⅰ)和核酸外切酶Ⅶ(Exo Ⅶ)属于单链的核酸外切酶,而双链的核酸外切酶则包括了大肠杆菌核酸外切酶Ⅲ(Exo Ⅲ)、λ噬菌体核酸外切酶(λ exo)以及T7核酸外切酶(T7 exo)等。尽管不像核酸内切酶那样具有对催化水解底物存在较强的碱基序列依赖性,不同的核酸外切酶存在不同的水解条件,可以区别出从分子链的3′末端或5′末端开始切断和对单链DNA或双链DNA具有特异性作用。为了更好地理解和应用核酸外切酶辅助的信号放大策略,本文总结了在生物分析研究中应用较多的几种核酸外切酶,如表1和图1所示。以核酸外切酶Ⅲ为例,该酶的切割底物为双链DNA,切割方向为3′→5′,且要求3′端为平齐或者凹陷末端,切割后产物为5′-单核苷酸。
3 核酸外切酶辅助的信号放大策略在生化分析中的应用
3.1 核酸检测
核酸是组成生命体的最主要生物大分子之一,是细胞内携带遗传信息的物质,它控制着蛋白质的合成和有机体细胞的机能,在生物的生长、发育、繁殖、遗传和变异等生命活动中占有极其重要的地位[16~18]。核酸序列中如果出现一个或几个核苷酸的取代、缺失或插入等微小的改变,都将引起基因突变或多态性,导致遗传病或其他疾病的出现[19,20]。因此,对人体的血液、组织及体液等样品中特定基因序列和与遗传疾病相关的单碱基突变的检测分析,在分子生物学、疾病早期诊断与治疗、发病机理研究以及药物筛选等方面都具有重大意义[21~25]。
最早的核酸检测方法是基于核酸杂交原理的分析方法,主要包括有Southern印迹杂交、Northern印迹杂交和原位杂交等[26~28]。然而当靶标序列浓度很低时,这种直接杂交技术的检测效果比较差,灵敏度低,大大限制了其广泛应用。随着生物技术的不断发展,一些新型的放大检测技术不断涌现。如体外扩增技术[29~31]、酶联催化反应[32,33]、脱氧核酶催化[34,35]、核酸酶辅助等[36~39]。其中,核酸外切酶辅助的靶标循环技术应用尤为广泛[40~44]。
2010年,Zuo等[41]使用分子信标作为信号探针,发展了一种核酸外切酶Ⅲ辅助的靶标循环信号放大技术用于DNA的高灵敏检测。实验原理如图2所示,在分子信标的5′端标记荧光团,与5′端平齐的3′端中间位置标记淬灭基团(分子信标3′端有7个碱基的突出序列用于抵抗核酸外切酶Ⅲ的水解),形成发夹后,荧光基团和淬灭基团距离非常近,从而使荧光基团的荧光被淬灭。当目标DNA存在时,分子信标被打开形成能被核酸外切酶Ⅲ水解的3′平末端,核酸外切酶Ⅲ沿双链的3′→5′方向逐步切除单核苷酸,并释放出荧光基团。而同时释放的目标DNA能与另一个分子信标杂交,引起酶切循环信号放大,这种方法对DNA的检出限达到20 amol/L。
相比昂贵的核酸探针标记,免标记的方法更简便、廉价。基于类似的原理,Xu等[42]利用λ核酸外切酶辅助的信号放大作用设计了一种超灵敏的电化学传感器。通过铁氰根离子在靶标存在与否情况下与电极的远近,表现出不同的电化学阻抗来特异性检测乳腺癌基因BRCA1。此外,Kong等[43]将核酸外切酶辅助的靶标循环与金纳米颗粒联用,实现双重放大免标记检测DNA,该方法检出限可以达到0.6 pmol/L。
除了对样品中DNA的检测外,RNA尤其是microRNA (miRNA)(被视为一种新型的肿瘤标志物)的检测也非常重要。Wang等[44]利用电化学方法,结合T7核酸外切酶的循环放大作用,设计了一种超灵敏检测miRNA的方法。其原理如图3所示:首先将能与miRNA互补的DNA探针的3′端修饰于金电极表面,此时由于表面覆盖有大量DNA使得阻抗信号较大。当加入miRNA之后,miRNA与DNA探针形成双链,在T7核酸外切酶的作用下,修饰在金电极上的DNA探针从5′端开始被水解,同时释放出的靶标miRNA能与下一个捕获探针杂交,实现循环放大。最后,金电极表面的DNA探针被剪切掉,导致电化学阻抗信号降低,进而实现miRNA的高灵敏检测。
图3 基于T7核酸外切酶辅助的信号循环放大方法检测miRNA的原理图[44]
Fig.3 Schematic illustration of T7 exo-assisted signal amplification system for miRNA detection[44]
此外,核酸序列中如果出现一个或几个核苷酸的改变,都将引起基因突变,导致遗传或其它疾病的出现。其中单核苷酸多态性(Single nucleotide polymorphisms, SNPs)占所有多态性的90%以上,是频率最高、信息含量最多的一种变异,也是最多见的遗传变异类型[45,46], 如镰刀形红细胞贫血、β-地中海贫血、囊性纤维化病以及肿瘤等[47,48]。由于单核苷酸多态性通常只是核酸序列中一个核苷酸改变,类似非靶标核酸的存在,因此针对核酸检测的一些方法也被用于与遗传疾病相关度较高的单碱基突变的检测分析。2014年,Wu等[49]利用分别标有荧光基团和淬灭基团分子探针来特异性检测单核苷酸多态性。最近,Wu等[50]为了避免分子探针双标效率低,费用昂贵等缺点,结合金颗粒和核酸外切酶Ⅲ设计了一种比色方法, 用于区分单碱基错配。
3.2 蛋白质检测
蛋白质是基因表达的最终产物,是细胞内各类代谢和调控等生命功能的执行者,也是致病因子、药物对机体作用的最重要的靶标分子。人类的绝大多数疾病都是由蛋白质表达异常引起的,蛋白的表达指示了细胞的生理和病理状态,揭示了药物及环境因素对生命体的影响。因此,系统性的蛋白质研究和分析对理解生命现象、监测疾病进程和药物作用等方面都具有非常重要的意义[51,52]。
基于传统的免疫分析是常规的蛋白质检测方法,通常在抗原或抗体上标记放射性物质、酶或荧光素等,然后利用抗原-抗体间的特异性反应来实现对蛋白质的灵敏特异性检测[53~59]。虽然免疫分析可以对蛋白质实现高灵敏度检测,但以上技术实验过程复杂,操作繁琐,效率低,不适合系统化、大规模的蛋白质组学研究,且功能化修饰可能影响抗原抗体间的亲和力。因此,发展一些简单、快速、灵敏的蛋白质分析新方法显得尤为重要。
近年来,基于蛋白与核酸的作用以及核酸与工具酶的作用,发展了一系列新型的蛋白质分析方法[60~64]。这些方法不仅避免了传统蛋白质检测方法中操作复杂,常需对蛋白质进行标记,不易保持其生物活性等缺点,还有效地提高了蛋白质检测的效率与灵敏度。2010年,Ou等[60]基于TATA结合蛋白(TBP)与含有TATA序列的DNA的特异结合能有效抵抗核酸外切酶的作用,设计了一种核酸外切酶Ⅲ辅助的信号放大比色检测蛋白质的新方法。如图4所示,首先在不同的金纳米颗粒上分别修饰上能互补的单链DNA,当两种金纳米颗粒按一定比例混合后,互补DNA之间的碱基作用力使得金纳米颗粒相互靠近,从而产生金纳米颗粒团聚效应,肉眼看到金纳米颗粒溶液由酒红色变成暗紫色。然后,加入核酸外切酶Ⅲ,当TBP存在时,因TBP与含有TATA序列DNA特异性结合,能对DNA 3′端有保护作用(空间位阻效应),使得核酸外切酶Ⅲ不能切割DNA链,从而金颗粒溶液一直呈暗紫色;当TBP不存在时,DNA链失去蛋白的保护作用,核酸外切酶Ⅲ从DNA 3′端切割,使得金颗粒彼此远离,溶液呈酒红色,从而实现对DNA特异结合蛋白TBP的比色检测。
图4 基于核酸外切酶Ⅲ辅助的信号循环放大方法和金颗粒检测DNA特异结合蛋白的原理图[60]
Fig.4 Schematic illustration of Exo Ⅲ-assisted signal amplification system and gold nanoparticles for sequence-specific DNA-binding protein detection[60]
随着更多的靶标蛋白的核酸适配体成功筛选,基于适配体作为识别元件的核酸外切酶辅助信号放大的蛋白质检测也备受关注。Chen等[61]发明了一种基于核酸适配体和核酸外切酶Ⅲ辅助下循环放大的平台用于检测溶菌酶。实验原理如图5所示,设计一个发夹结构DNA,DNA链的一部分为溶菌酶的核酸适配体。当有溶菌酶存在的情况下,探针发生空间构象变化使得非核酸适配体部分暴露出,然后加入标记了荧光基团的信号探针,该探针与非核酸适配体部分互补配对,形成平齐末端的DNA双链,在核酸外切酶Ⅲ的作用下,信号探针被剪切,释放出荧光基团,被打开的发夹探针可以循环利用,释放出大量荧光基团,由于荧光分子无法被吸附到氧化石墨烯表面,从而呈现出强的荧光信号。当不存在溶菌酶时,发夹结构无法打开,信号探针无法与其互补结合,核酸外切酶Ⅲ也无法切割,荧光基团一直被标记在信号探针上,信号探针与氧化石墨烯结合,荧光基团被氧化石墨烯淬灭,呈现很低的荧光信号。
基于类似的原理,凝血酶与其对应的核酸适配体结合后构型会发生变化,Wang等[62]设计了一种检测凝血酶的方法,该方法特异性好,灵敏度高,可达到对几个蛋白分子的准确定量。此外,利用一些核酸外切酶对底物的偏好性(如磷酸化、甲基化等),发展了一些磷酸激酶、磷酸化酶、甲基转移酶等的检测新方法。Liu等[63]利用λ核酸外切酶对底物5′磷酸化的要求,结合氧化石墨烯平台设计了一种快速检测T4多核苷酸激酶活性的方法。
图5 基于氧化石墨烯和核酸外切酶Ⅲ辅助的信号循环放大方法检测溶菌酶的原理图[61]
Fig.5 Schematic illustration of GO and Exo Ⅲ-assisted signal amplification system for lysozyme detection[61]
3.3 生物小分子检测
除了核酸、蛋白质等生物大分子外,一些生物活性小分子在机体的新陈代谢及生命过程中也起着重要作用,与人类的健康息息相关。如三磷酸腺苷(ATP)发生水解时,能产生大量供给人体的日常所需的生命活动的能量;还原型的谷胱甘肽(GSH)可以通过与过多的自由基或者对身体有害处的重金属等物质结合,以达到中和有毒物质的作用,并能够将之排出体外,所以GSH是人体内部特别的抗氧化剂跟自由基清除剂。以GSH、ATP等为代表的细胞内小分子活性物质的检测对研究机体的生理功能和疾病早期诊断具有重要意义。目前,用于生物活性分子的分析检测手段主要有荧光、比色、电化学以及同位素标记等。其中,利用荧光方法分析小分子具有灵敏度高、分析快捷、样品制备简单等优点而被广泛关注。随着配体指数富集系统进化技术(SELEX)的发展,一些小分子的核酸适配体(Aptamer)被发现,利用其适配体与小分子的特异性结合,已发展了许多新的小分子荧光分析新方法[65~67]。工具酶对作用底物的结构有较强的依赖性,而这些小分子与核酸适配体的结合又能诱导核酸适配体构型的变化,因而,一些工具酶也被广泛用于小分子的放大检测[68~71]。
如图6所示,Xu等[68]设计了一种基于核酸外切酶Ⅲ催化的目标物循环放大方法免标记检测ATP。利用ATP的核酸适配体构建了一个发夹探针,且使得茎部3′端突出,以避免被核酸外切酶Ⅲ降解。当有ATP存在时,ATP与发夹探针中的核酸适配体序列结合,使得核酸适配体的构型发生变化,导致茎部3′端凹陷,加入核酸外切酶Ⅲ后即可从3′端开始剪切双链部分,最终形成单链DNA,并释放出ATP,而ATP即可继续循环利用,实现循环放大,双链特异性染料无法嵌入循环产生的单链DNA,因此没有荧光信号。而没有目标ATP存在时,发夹探针的茎部双链部分能嵌入SYBR Green I呈现出非常高的荧光信号。
Zheng等[69]利用核酸适配体与靶标结合产生的空间位阻作用达到抗酶切的效果,设计了一种基于核酸外切酶Ⅰ辅助的分析检测平台,可用于离子、小分子物质及蛋白的检测。
图6 基于核酸外切酶Ⅲ辅助的目标物循环放大方法检测ATP的原理图[68]
Fig.6 Schematic illustration of Exo Ⅲ-assisted target amplification system for ATP detection[68]
3.4 离子检测
重金属离子Hg2+和Ag2+由于其性质相对比较稳定,在环境中很难被生物降解,且生物毒性高,因此,重金属污染严重危害环境及生物的生存。由于重金属污染以及重金属随着食物链的富集作用,使得重金属离子广泛存在于水、土壤和食物当中,
图7 基于核酸外切酶Ⅲ辅助的信号放大方法检测Hg2+的原理图[80]
Fig.7 Schematic illustration of Exo Ⅲ-assisted signal amplification system for Hg2+ detection[80]
然而重金属的摄入会直接损害人体健康,过量甚至会危及生命[72,73],因此针对重金属离子的监测显得极为重要。传统的检测方法是基于专业仪器的原子吸收光谱、原子发射光谱以及电感耦合等离子体质谱法等,这些方法存在操作复杂、费时费力、成本高等缺点。近些年,一些基于小分子发色团、纳米材料、核酶以及聚合物材料的重金属离子传感检测方法被不断发展[74~76]。同时,基于DNA的特殊碱基或位点能特异性地与某些金属离子结合,形成非自然碱基对(即金属离子稳定的非沃森-克里克碱基配对系统,如“T-Hg2+-T”、“C-Ag+-C”),许多针对相应离子的传感器被发展出来[77~79]。正是由于这些离子与核酸的结合,许多工具酶,特别是核酸外切酶也被引入相应离子的放大检测体系[80,81]。如Chen等[80]发明了一种基于核酸外切酶Ⅲ辅助的信号放大方法可视化检测Hg2+的一次性条带生物传感器,其原理如图7所示,发夹DNA和辅助DNA中间部分可互补配对,其中发夹DNA的3′端和辅助DNA的5′均为富T序列,没有Hg2+存在时,二者无法杂交以打开发夹DNA;只有当Hg2+存在时,通过T-Hg2+-T非自然配对使得形成具有平齐末端的双链DNA,进而被核酸外切酶Ⅲ识别并从3′端开始剪切,释放出的辅助DNA和Hg2+继续循环利用,产生大量ssDNA。在B部分中,结合部分(Conjugate pad)含有大量修饰了DNA的金颗粒(AuNPs-DNA probe1),检测条带区域(TZ)处含有修饰了链霉亲和素和生物素的DNA(SA-biotin-DNA probe2),对照条带区域(CZ)处含有修饰了链霉亲和素和生物素的另一条DNA(SA-biotin-DNA probe3)。其中AuNPs-DNA probe1与SA-biotin-DNA probe3是互补的,只要条带部分完好,金颗粒被富集于CZ条带处显色,该条带可以检验条带传感器是否完好。当检测体系中有Hg2+存在时,产生的ssDNA可以与AuNPs-DNA probe1和SA-biotin-DNA probe2杂交,从而使金颗粒富集于TZ条带处显色;而当没有Hg2+存在时,无法产生ssDNA,因此TZ条带处不能显色。该方法方便快捷,可直接肉眼观察到结果,且灵敏度高,检出限可达1 pmol/L。类似T-Hg2+-T非自然碱基配对,基于C-Ag+-C效应和核酸外切酶Ⅲ辅助放大,Xu等[79]设计了一种的循环放大方法检测Ag+,其检出限可达0.03 nmol/L。
4 展 望
核酸外切酶在生化分析领域的应用发展极其快速而且应用方式变化层出不穷。纵观核酸外切酶在生化分析中的应用情况,其在生化分析中的应用发展未来将有可能呈现出以下趋势,包括:(1)多元化信号获取方式。可以通过改进仪器达到高灵敏的检测(如荧光信号、电化学信号等),也可通过肉眼直接观察的比色方法以及试纸条显色等方法得出结果,未来可能有更多、更灵敏的信号输出方式;(2)酶切循环放大和功能化纳米材料信号放大相结合实现多重放大。例如酶和金属纳米颗粒结合应用,先通过酶切实现信号放大,然后通过结合金属纳米颗粒二级放大实现更灵敏的检测; (3)向生化传感的实用性方向发展。利用核酸外切酶来实现信号放大的传感器通常都比较轻巧,不需要配备严格的实验条件和贵重的仪器,可用于发展便携的传感器或检测试纸条等,因此这类传感器有望实现即时检测(POCT)和临床应用;(4)复杂生物样品的检测。基于核酸外切酶的放大检测方法,目前还主要应用于体外缓冲液样品的检测,随着更多的离子、分子、细胞等与核酸结合的发现,核酸外切酶也将用于更多样性的分析物的检测,其难点和挑战是在更为复杂的生物样品(如体液、细胞等)中实现目标物的检测。我们相信,随着对核酸外切酶性质和应用的进一步探索,核酸外切酶在生化分析领域将会有更加广阔的应用前景。
References
1 Gerasimova Y V, Kolpashchikov D M. Chem. Soc. Rev., 2014, 43(17): 6405-6438
2 Minunni M, Tombelli S, Fonti J, Spiriti M M, Mascini M, Bogani P, Buiatti M. J. Am. Chem. Soc., 2005, 127(22): 7966-7967
3 Banér J, Nilsson M, Mendel-Hartvig M, Landegren U. Nucleic Acids Res., 1998, 26(22): 5073-5078
4 Tao C, Yan Y, Xiang H, Zhu D, Cheng W, Ju H, Ding S. Chem. Commun., 2015, 51(20): 4220-4222
5 Wu Z K, Zhou D M, Wu Z, Chu X, Yu R Q, Jiang J H.Chem. Commun., 2015, 51(14): 2954-2956
6 Sun J, Jiang W, Zhu J, Li W, Wang L. Biosens. Bioelectron., 2015, 70: 15-20
7 Bao T, Wen W, Zhang X, Xia Q, Wang S. Biosens. Bioelectron., 2015, 70: 318-323
8 Liu S, Liu T, Wang L.Chem. Commun., 2015, 51(1): 176-179
9 Liu X, Li W, Hou T, Dong S, Yu G, Li F. Anal. Chem., 2015, 87(7): 4030-4036
10 Guo X, Liu P, Yang X, Wang K, Wang Q, Guo Q, Huang J, Liu J, Song C, Li W. Analyst, 2015, 140(6): 2016-2022
11 Guo X, Yang X, Liu P, Wang K, Wang Q, Guo Q, Huang J, Li W, Xu F, Song C. Analyst, 2015, 140(6): 4291-4297
12 Liu P, Sun S, Guo X, Yang X, Huang J, Wang K, Wang Q, Liu J, He L. Anal. Chem., 2015, 87(5): 2665-2671
13 Miyake Y, Togashi H, Tashiro M, Yamaguchi H, Oda S, Kudo M Y, Kondo Y, Sawa R, Fujimoto T, Machinami T, Ono A. J. Am. Chem. Soc., 2006, 128(7): 2172-2173
14 Liu Y , Luo M, Xiang X, Chen C, Ji X, Chen L, He Z. Chem. Commun., 2014, 50(20): 2679-2681
15 GUO Qiu-Ping, ZHAO Xia-Yu, XIE Qin, WANG Ke-Min, WAN Jun, YUAN Bao-Yin, TAN Yu-Yu. Chem. J. Chinese Universities, 2014, 35(8): 1646-1651
郭秋平, 赵下雨, 谢 琴, 王柯敏, 万 俊, 袁宝银, 谭誉宇. 高等学校化学学报, 2014, 35(8): 1646-1651
16 Woychik R P, Klebig M L, Justice M J, Magnuson T R, Avrer E D. Mutat. Res-Fund Mol. M., 1998, 400(1): 3-14
17 Williams G T, Smith C A. J. Cell Sci., 1993, 74(5): 777-779
18 ZHONG Bi-Ling,SONG Yong-Sheng. Chin. J. Cancer., 2000, 19(5): 432-435
钟碧玲, 宋永生. 中国癌症杂志, 2000, 19(5): 432-435
19 ZHENG Qin, LIU Hua, ZENG Ting-Ting, YE Yuan-Xin,JIANG Neng-Gang,SU Jun. Chinese Journal of Evidence-based Medicine, 2013, 13(2): 170-175
郑 沁, 刘 华, 曾婷婷, 叶远馨, 蒋能刚, 粟 军. 中国循证医学杂志, 2013, 13(2): 170-175
20 ZHAO Hong-Shan, HUANG Shang-Zhi. Chinese J Med Genet., 2000, 17(1): 6-9
赵红珊, 黄尚志. 中华医学遗传学杂志, 2000, 17(1): 6-9
21 Osada M, Ohba M, Kawahara C, Ishioka C, Kanamaru R, Katoh I, Katoh I, kawa Y, Nimura Y, N akagawara A, Obinata M I, Kawa S. Nat. Med., 1998, 4(7): 839-843
22 Shi S T, Yang G Y, Wang L D, Xue Z, Feng B, Ding W, Xing E P, Yang C S. Carcinogenesis, 1999, 20(4): 591-597
23 CAI Wei-Ming, HU Min-Jun. Zhejiang Journal of Integrated Traditional Chinese and Western Medicine, 2002, 12(11): 661-666
蔡卫民, 胡敏君. 浙江中西医结合杂志, 2002, 12(11): 661-666
24 ZENG Yi-Tao. Chinese J Hematol., 1996, 17(8): 393-394
曾溢滔. 中华血液学杂志, 1996, 17(8): 393-394
25 Marton M J, DeRisi J L, Bennett H A, Iyer V R, Meyer M R, Roberts C J, Stoughton R, Burchard J, Slade D, Dai H Y, Bassett Jr D E, Hartwel L H, Brown, P O, Friend, S H. Nat. Med., 1998, 4(11): 1293-1301
26 Mulbry W W, Karns J S, Kearney P C, Nelson J O, McDaniel C S, Wild J. R. Appl Environ Microb., 1986, 51(5): 926-930
27 Correa R R, Mariash C N, Rosenberg M E. Biotechniques., 1992, 12(2): 154-158
28 Yang H, McLeese J, Weisbart M, Dionne J L, Lemaire I, Aubin R A.Nucleic Acids Res., 1993, 21(14): 3337-3338
29 Krokene P, Barnes I, Wingfield B D, Wingfield M J. Mycologia, 2004, 96(6): 1352-1354
30 Odin E, Larsson L, Aram M, Gustavsson B, Larsson P A. Tumor Biol., 1998, 19(3): 167-175
31 Pfaffl M W.Nucleic Acids Res., 2001, 29(9): e45-e45
32 De La Rica R, Stevens M M. Nat. Nanotechnol., 2012, 7(12): 821-824
33 Wang Q, Liu F, Yang X H, Wang K M, Liu P, Liu J B, Huang J, Wang H. Sensor Actuat B-Chem., 2013, 186: 515-520
34 Deng M, Zhang D, Zhou, Y, Zhou X. J. Am. Chem. Soc., 2008, 130(39): 13095-13102
35 Zhao X, Gong L, Zhang X B, Yang B, Fu T, Hu R, Tan W H ,Yu R Q. Anal. Chem., 2013, 85(7): 3614-3620
36 Yin B C, Liu Y Q, Ye B C.J. Am. Chem. Soc., 2012, 134(11): 5064-5067
37 Bi S, Zhang J, Zhang S.Chem. Commun., 2010, 46(30): 5509-5511
38 Li W, Wu P, Zhang H, Cai C. Anal. Chem., 2012, 84(17): 7583-7590
39 Ren W, Gao Z F, Li N B, Luo H Q. Biosens. Bioelectron., 2015, 63: 153-158
40 Guo Q, Yang X, Wang K, Tan W, Li W, Tang H, Li H. Nucleic Acids Res., 2009, gkn1024
41 Zuo X, Xia F, Xiao Y, Plaxco K W.J. Am. Chem. Soc., 2010, 132(6): 1816-1818
42 Song C, Yang X, Wang K, Wang Q, Liu J, Huang J, He L, Liu P, Qing Z H, Liu W. Chem. Commun., 2015, 51(10): 1815-1818
43 Kong R M, Song Z L, Meng H M, Zhang X B, Shen G L, Yu R Q. Biosens. Bioelectron., 2014, 54: 442-447
44 Wang M, Fu Z, Li B, Zhou Y, Yin H, Ai S. Anal. Chem., 2014, 86(12): 5606-5610
45 Risch N, Merikangas K. Science, 1996, 273(5281): 1516-1517
46 Kruglyak L. Nat Genet., 1997, 17(1): 21-24
47 WU Guan-Yun, WANG Shen-Wu. Gene Diagnosis. Beijing: People′s Medical Publishing House, 1988: 131-142
吴冠芸, 王申五. 基因诊断. 北京: 人民卫生出版社, 1988: 131-142
48 WU Guan-Yun, FANG Fu-De. Gene Diagnosis Technology and Application. Beijing: Beijing Medical University and China Xie-He. Medical University Joint Publishing House, 1992: c176-177
吴冠芸, 方福德. 基因诊断技术及应用, 北京: 北京医科大学、北京协和医科大学联合出版社, 1992: c176-177
49 Wu T, Xiao X, Zhang Z, Zhao M. Chem. Sci., 2015, 6(2): 1206-1211
50 Wu S, Liang P, Yu H, Xu X, Liu Y, Lou X, Xiao Y. Anal. Chem., 2014, 86(7): 3461-3467
51 Mikami T, Yanagisawa N, Baba H, Koike M, Okayasu I. Cancer, 1999, 85(2): 318-325
52 JING Li, ZHANG Jian-Zhong, GUO Feng-Ying. Chin. J. Cancer., 2003, 13(1): 27-29
景 丽, 张建中, 郭凤英. 中国癌症杂志, 2003, 13(1): 27-29
53 Ellington A D, Szostak J W. Nature, 1990, 346(6287): 818-822
54 Tuerk C, Gold L. Science, 1990, 249(4968): 505-510
55 Guo W M, Kong K W, Brown C J, Quah S T, Yeo H L, Hoon S, Seow Y.Mol. Ther-Nucl. acids., 2014, 3(12): e217
56 Famulok M, Hartig J S, Mayer G. Chem. Rev., 2007, 107(9): 3715-3743
57 Voller A, Bartlett A, Bidwell D E. J. Clin. Pathol., 1978, 31(6): 507-520
58 Blake M S, Johnston K H, Russell-Jones G J, Gotschlich E C. Anal. Biochem., 1984, 136(1): 175-179
59 Schaeffer M, Orsi E V, Wide lock D. Bact. Rev., 1964, 28(4): 402-408
60 Ou L J, Jin P Y, Chu X, Jiang J H, Yu R Q. Anal. Chem., 2010, 82(14): 6015-6024
61 Chen C, Zhao J, Jiang J, Yu R. Talanta, 2012, 101: 357-361
62 Wang X L, Li F, Su Y H, Sun X, Li X B, Schluesener H J, Tang F, Xu S Q. Anal. Chem., 2004, 76(19): 5605-5610
63 Liu L, Lei J, Gao F, Ju H X. Talanta, 2013, 115: 819-822
64 Li W, Liu X, Hou T, Li H, Li F., Biosens. Bioelectron., 2015, 70: 304-309
65 Kim Y S, Kim J H, Kim I A, Lee S J, Jurng J, Gu M B. Biosens. Bioelectron., 2010, 26(4): 1644-1649
66 HyunáKim T. Lab Chip, 2011, 11(1): 52-56
67 Deng C, Chen J, Nie L, Nie Z, Yao S. Anal. Chem., 2009, 81(24): 9972-9978
68 Xu Y, Xu J, Xiang Y, Yuan R, Chai Y. Biosens. Bioelectron., 2014, 51: 293-296
69 Zheng D, Zou R, Lou X. Anal. Chem., 2012, 84(8): 3554-3560
70 Wei Y, Chen Y, Li H, Shuang S, Dong C, Wang G. Biosens. Bioelectron., 2015, 63: 311-316
71 Sun J, Jiang W, Zhu J, Li W, Wang L. Biosens. Bioelectron., 2015, 70: 15-20
72 Renzoni A, Zino F, Franchi E. Environ. Res., 1998, 77(2): 68-72
73 Nichols J W, Brown S, Wood C M, Walsh P J, Playle R C. Aquat. Toxicol., 2006, 78(3): 253-261
74 Guo X, Qian X, Jia L. J. Am. Chem. Soc., 2004, 126(8): 2272-2273
75 Xue X, Wang F, Liu X. J. Am. Chem. Soc., 2008, 130(11): 3244-3245
76 Ono A, Togashi H. Angew. Chem. Int. Ed., 2004, 43(33): 4300-4302
77 Yin J, He X, Jia X, Wang K, Xu F. Analyst, 2013, 138(8): 2350-2356
78 Zhao C, Qu K, Song Y, Xu C, Ren J, Qu X. Chem-Eur J., 2010, 16(27): 8147-8154
79 Xu G, Wang G, He X, Zhu Y, Chen L, Zhang X. Analyst, 2013, 138(22): 6900-6906
80 Chen J, Zhou S, Wen J. Anal. Chem., 2014, 86(6): 3108-3114
81 Xuan F, Luo X, Hsing I M. Anal. Chem., 2013, 85(9): 4586-4593

