胡倩梅 杨会会 朱华玉 胡建斌 杨路明


摘? ? 要: 为寻找调控果面茸毛、果面沟以及果面瘤表型变异的位点,调查200份甜瓜种质在2017年和2018年的表型,并对这200份种质进行全基因组重测序,利用全基因組关联分析的方法对其进行关联。结果在3号染色体上找到了一个与果面茸毛显著相关的SNP;在11号染色体上找到了2个与果面沟显著相关的SNP,在5号、7号和11号染色体上各找到了一个与果面沟深显著相关的SNP;在12条染色体上均找到了与果面瘤显著相关的SNP,所有与果面瘤显著相关的SNP标记中有8个标记显著性远远高于其他标记。进一步对果面茸毛的1个显著SNP,果面沟的5个显著SNP和果面瘤的8个SNP进行了基因注释,共计找到了146个基因。研究结果有助于甜瓜果面茸毛、果面瘤以及果面沟性状的快速精细定位,也将为甜瓜果实遗传改良提供重要参考依据。
关键词: 甜瓜;果面茸毛;果面沟;果面瘤;全基因组关联分析
Genome-wide association analysis of fruit fluff, fruit surface tumor and fruit surface ditch in melon
HU Qianmei1, YANG Huihui1, ZHU Huayu1,2, HU Jianbin1,2, YANG Luming1,2
(1.College of Horticulture, Henan Agricultural University, Zhengzhou 450002, Henan, China; 2. Henan Key Laboratory of Fruit and Cucurbit Biology, Zhengzhou 450002, Henan, China)
Abstract: Based on the phenotyping and re-sequencing of 200 melon germplasms, the genome-wide association analysis was used to identify loci associated with fruit villi, fruit sulcus, and fruit surface tumors in melon. These traits were investigated of 200 melon germplasms in 2017 and 2018, and genome-wide resequencing of these 200 germplasms was conducted for SNP detection. As a result, 1 SNP was found on chromosome 3 that was significantly associated with fruit fluff. 2 SNPs were found on chromosome 11 that were significantly associated with the fruit surface ditch. There was only 1 SNP was found significantly associated with the fruit surface ditch on the chromosome 5, 7 and 11, respectively. SNPs that were significantly associated with fruit surface tumors were found on all 12 chromosomes. Among of them, 8 SNPs had much higher significantly associated than the other SNPs, therefore those SNPs were used in further study. Totally 14 SNPs were detected associated for fruit villi, fruit sulcus, and fruit surface tumors, and 146 genes flanking these SNP were annotated. The SNPs significantly associated withthese traits will be helpful to quickly and finely locate candidate genes, and will also provide an important reference for the genetic improvement of melon fruit.
Key words: Melon; Fruit fluff; Fruit groove; Fruit surface tumor; Genome-wide association analysis
甜瓜(Cucumis melo L.)为葫芦科(Cucurbitaceae)作物,染色体数为2n=2x=24。甜瓜属于喜温、喜光植物,在28~32 ℃范围内可以正常生长,低于13 ℃即停止生长[1]。甜瓜营养物质含量丰富,是世界十大水果之一。甜瓜自身的表型多样性极其丰富,不同类型的甜瓜在果面茸毛、果面瘤、果面沟、果皮颜色、果肉颜色、瓜瓤颜色、种子颜色、果面网纹、可溶性固形物含量、叶片形状等诸多方面有很大的差异,其多样性在葫芦科植物中仅次于南瓜。2012年甜瓜全基因组测序结果公布,基因组大小为454 Mb,预测共有27 427个基因[2],为开展甜瓜分子育种研究提供了极大的便利。在过去的十几年间大量的研究工作者对甜瓜的果皮颜色、果面覆纹、果肉颜色、果肉含糖量、果实质量、果实长度、果实宽度、果肉厚度、叶面积、叶片缺刻、种子宽度、种子长度以及种子质量[3-14]等农艺性状做了深入研究,然而对成熟果实的茸毛自然脱落、果面瘤和果面沟所做的研究还相对较少。
果面茸毛是指果实成熟后果皮表面的茸毛,与先前Palomares-Rius等[15]所研究的TypeⅠ型茸毛以及Zhu等[16]所研究的无毛突变体均不同。果面瘤是指成熟果实表面的瘤状物凸起,先前研究中多認为该性状是由环境引起的,但笔者在研究中发现部分材料较易产生果面瘤,而部分材料则不易产生果面瘤;果面沟指成熟果实表面的凹陷,我们一般将果面沟分为果面沟有无、果面沟浅和果面沟深这3个性状,果面沟影响甜瓜的果皮结构、果实受力以及裂果,王学征等[17]通过构建6世代定位到1个与果面沟形成相关的基因。
全基因组关联分析( GWAS)是指在全基因组范围内,通过高密度的SNP标记挖掘控制表型性状变异基因座的统计分析方法。关联分析的方法最早应用于人类相关疾病case-control研究中[18],受人类疾病遗传学研究的启示,GWAS也被运用到作物遗传和育种研究中并取得重要进展[19-20],本研究通过对200份甜瓜种质进行重测序,利用 GWAS的方法对成熟果实果面茸毛的脱落、果面瘤、果面沟有无、果面沟浅以及果面沟深这5个性状进行研究,以期能够找到真正造成表型变异的突变位点。
1 材料与方法
1.1 材料
供试材料来自于从世界各地收集的200份甜瓜种质,包括88份厚皮种质,91份薄皮种质和21份野生种质,其中11份种质由胡建斌收集,77份种质来源于美国农业部,112份种质来自于中国农业科学院郑州果树所。
1.2 方法
1.2.1 田间试验 本研究田间试验于2017年、2018年在河南农业大学科教园区毛庄试验基地的塑料大棚进行。2月中旬进行温汤浸种催芽,材料播种于72孔穴盘,在日光温室中进行育苗。所有材料3叶1心时进行定植,每年设置2个重复,每个重复内每份材料定植4个单株。田间水、肥以及整枝按照常规管理。所有材料于果实成熟时期进行性状调查和统计,调查标准参考《甜瓜种质资源描述规范和数据标准》[21]的标准与方法进行统计、分级和赋值。
1.2.2 测序样品制备 植株定植30 d后,采用混合采样法,每个材料混合采取4个单株的幼嫩叶片3~5 g,采用真空冷冻干燥器将叶片抽干,并用高通量组织粉碎机将叶片打磨成粉末状备用。提取总DNA,-80 ℃备用。
1.2.3 全基因组重测序 200份甜瓜种质的测序工作由北京百迈客生物科技有限公司完成,采用Illumina测序技术。测序完成后过滤掉带接头的reads、N含量超过10%的reads以及质量值低于10的碱基超过50%的reads。
1.2.4 SNP标记的检测与筛选 采用GATK(V4.0.9.0)call SNP[22],将双端read和单端read都作为 call SNP的初始reads数据。
1.2.5 群体遗传学分析 应用利用GCTA (V1.91.2)进行主成分分析,保留前3个特征向量,以其作为协变量放入全基因组关联分析模型中[23];利用GAPIT进行亲缘关系分析亲缘关系矩阵(K)[24]作为随机效应加入到全基因组关联分析模型中;LD值由PopLDdecay(V3.40)进行计算,用r?值来确定LD衰减距离。
1.2.6 全基因组关联分析 为减弱性状的复杂性对关联结果的影响,除去果面茸毛和果面瘤外,我们将果面沟分为果面沟有无、果面沟浅和果面沟深进行关联分析。本研究利用GAPIT中的一般线性模型(GLM)、混合线性模型(MLM)、压缩混合线性模型(CMLM)和多位点混合线性模型(MLMM)4个模型同时对5个性状进行关联,利用BH矫正进行筛选显著SNP,将同时在 5个模型中关联到的SNP标记进行后续分析。
1.2.7 SNP注释 根据公布的甜瓜基因组CM3.6.1_pseudomol.fa (V3.6.1)和注释文件CM4.0.gff3(V4.0)对获得的全基因组SNP进行注释。
2 结果与分析
2.1 全基因组重测序与SNP标记过滤
200份甜瓜种质的全基因组重测序共计获得1 849 Gb的clean测序数据,平均测序深度为22 X。通过GATK对SNP的过滤,共计获得2 989 591个SNP标记。
2.2 连锁不平衡分析
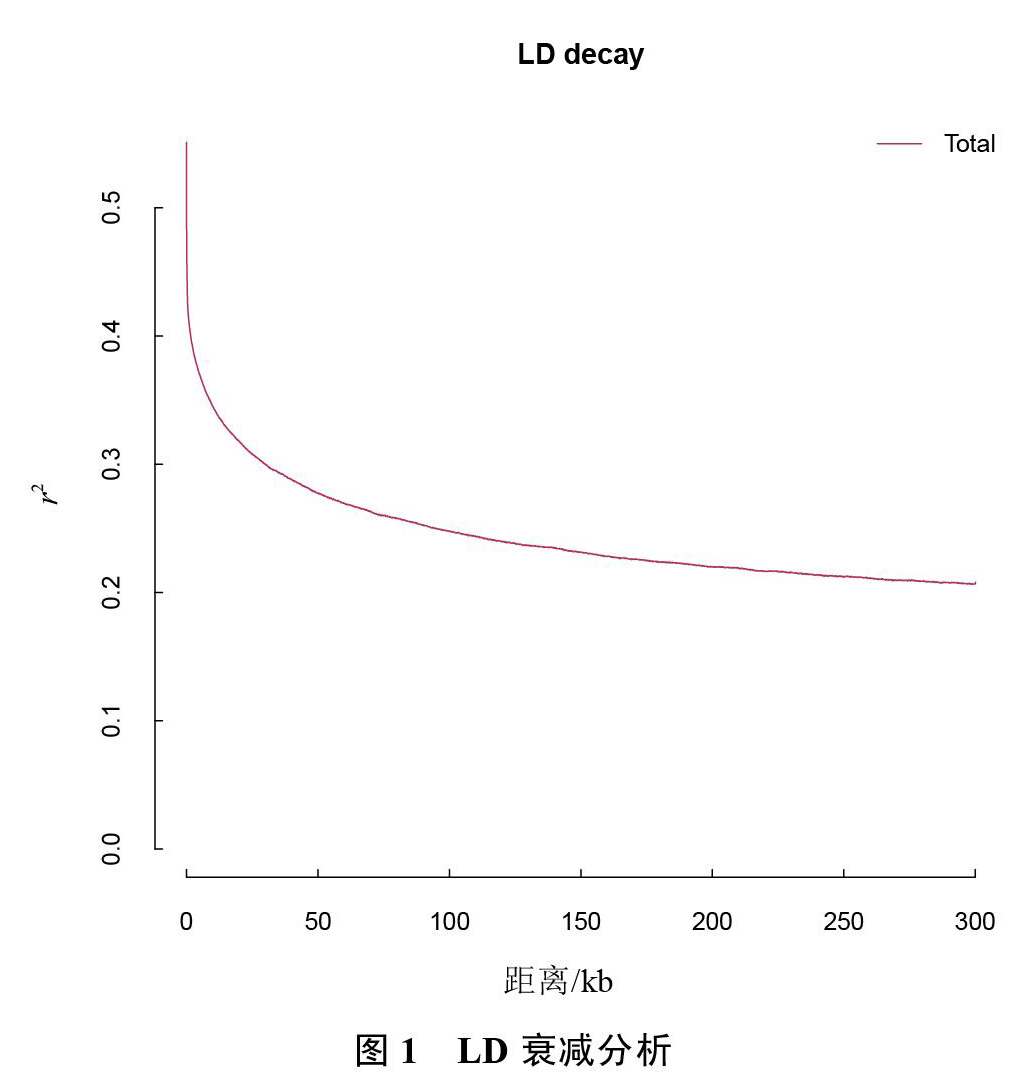
通过对200份种质材料进行LD衰减分析(图1),结果表明群体衰减距离为60 kb,将在检测到的显著SNP前后30 kb分析候选基因[25]。
2.3 全基因组关联分析
关联分析能够通过计算每个SNP位点与目标性状关联的可信值,从而对调控目标性状的位点进行预测,笔者通过对果面茸毛、果面瘤、果面沟有无、果面沟浅和果面沟深这5个农艺性状进行GLM、MLM、CMLM和MLMM等4种关联模型全基因组关联分析,发现果面茸毛有1个SNP(图2-A),位于3号染色体;果面瘤检测到了105个SNP(图2-B)在12条染色体均有分布;果面沟有无检测到了2个SNP(图2-C),位于11号染色体;果面沟浅没有检测到SNP(图2-D),果面沟深检测到3个SNP(图2-E),分别位于5号、7号和11号染色体。其中果面沟有无和果面沟深均检测到了标记chr11_24130439,果面瘤检测到的显著SNP中chr7_8087384、chr8_10804573、chr12_19247259、chr7_21474816、chr4_32540139、chr4_3278835、chr9_23361283和chr2_9400335这8个SNP的显著性最高,其P值分别为1.16E-53、1.80E-53、2.23E-47、1.96E-45、1.12E-37、6.33E-29、5.69E-24和1.16E-20,基于此对SNPs的注释中针对果面瘤我们着重研究这8个显著性最高的SNP(表1)。
2.4 SNP的注释
基于甜瓜基因组注释在14个SNP上下游区域30 kb测到146个基因,果面茸毛获得10个相关基因,果面沟有无获得14个基因,果面沟深获得33个基因,果面瘤獲得89个基因。经比对分析我们发现在果面瘤茸毛显著SNP标记chr3_256240260附近检测到的基因MELO3C011492.2和纤维素的分解、膜的组成以及纤维素酶的活性均有关联;与果面沟显著相关的SNP标记chr11_24130439附近检测到基因MELO3C019697.2和细胞的组成部分以及伸长相关,标记chr05_24592367附近检测到基因MELO3C004136.2能够调控细胞的单方向生长,标记chr07_2180656附近的基因MELO3C016777.2对分生组织的生长和纤维素微纤维组织均有调控作用;与果面瘤相关的SNP标记chr4_3278835附近的基因MELO3C003679.2和细胞质的组成成分相关。
3 讨 论
3.1 甜瓜全基因组关联分析
自2012年甜瓜基因组序列发表以来,利用全基因组关联分析的方法研究甜瓜相关性状的研究也相继发表,但其在甜瓜中的应用还比较薄弱,所用标记和关联的性状均较少。2015年齐振宇[9]利用14 689个SNP标记对构建的F2群体的6个性状进行全基因组关联分析,在1号和10号染色体检测到与节间长度相关的SNP位点,在1、4和8号染色体检测到与侧枝长相关联的SNP位点,在1、3、4、8、11和12号染色体检测到与叶面积相关的SNP位点,在10号染色体检测到与果皮颜色相关的SNP位点,在2号染色体上检测到与甜瓜抗白粉病相关的SNP。2016年Nimmakayala等[26]利用13 756个SNP标记通过GWAS和QTL连锁作图相互验证的方法将甜瓜果肉硬度定位到6、8、9、11和12号染色体。2017年王艳玲等[27]利用104个SSR标记对果实相关性状进行全基因组关联分析,将果实纵径、果实横径、果形指数、果肉厚度和果实鲜质量分别关联到10、5、9、8和10个SSR标记。本研究所用SNP标记数远远高于以上研究在甜瓜全基因组关联分析中所用的标记数,研究中所关联的性状还未发现有学者对其用全基因组关联分析的方法研究过,本研究丰富了甜瓜在全基因组关联分析方面的研究。
3.2 关联模型比较
Q-Q图可以通过判断图形中的点分布是否合理,进而推断出所用的统计模型是否合理。一般Q-Q图有两种比较合理的模型,第1种是观测值与期望值相同,第2种是Q-Q图的左下角期望值与观测值一致,右上角有少量位点高高翘起,超过期望值。第1种表明没有找到与性状显著相关的SNP,第2种是在进行关联分析中所期望得到的关联结果,左下角期望值与观测值一致表明了模型的合理性,右上角少量标记超出了期望值,表明对所关联性状关联到了与性状显著相关的SNP标记。笔者同时应用了4种关联模型,由本研究中的5个性状结果图可以看出一般线性模型和混合线性模型均有大量的标记观测值高于期望值,显然这是不符合生物学逻辑的。而压缩混合线性模型和多位点混合线性模型从Q-Q图上来看一方面比较符合生物学逻辑,另一方面这两个模型的一致性比较高,从侧面验证的检测到的显著性标记具有较高的可信度。
3.3 关联结果
果面茸毛、果面瘤以及果面沟是影响甜瓜外观品质和商业品质的重要因素,定位调控果面茸毛、果面瘤以及果面沟的基因位点可以揭示这3类性状的遗传机制,为利用分子育种技术培育外观品质和商业品质优异的新品种奠定基础。目前关于甜瓜茸毛的报道仅有2篇,Palomares-Rius等[15]所研究的Type I型茸毛主要表现为茸毛短小,且主要是对叶片的研究,Zhu等[16]所研究的无毛突变体在整个生育期整个植株上均表现为无毛,而本研究的果面茸毛仅在果实成熟期开始脱落,基于该性状对果实幼果的保护,是一个重要的农艺性状,目前尚且没有学者对其进行研究,笔者在200份甜瓜种质中关联到的位点(chr3_25624260)与前两位学者所关联到的位点(11号染色体和8号染色体)完全不同,说明这些性状是由不同位点控制的。王学征等[17]将果面沟QTL_qGroove-1-1定位到11号染色体末端的1.1 cM的区间内,这与本研究中关于果面沟所检测到11号染色体上的SNP标记比较接近,同时我们在5号的7号染色体发现了新的和果面沟显著相关的位点,可能果面沟并不是一个简单的质量性状,推测其可能是由多个位点进行调控的数量性状,这与王学征等[17]所认为的果面沟是由一对等位基因调控的结果所不同。甜瓜果面瘤的性状目前还没有学者进行研究。尽管本研究检测到了156个与表型显著相关的SNP,但没有对所检测到的SNP附近基因进行定量分析,无法确定所获得的标记附近基因对表型的调控模式,仍需后续研究对其进行深入分析研究。
参考文献
[1] 吴梅梅,张显,郑俊鶱,等.西瓜甜瓜抗寒性研究进展[J].中国瓜菜,2014,27(S1):1-5.
[2] GARCIA-MAS J,BENJAK A,SANSEVERINO W,et al.The genome of melon (Cucumis melo L.)[J].Proceedings of the National Academy of Sciences of the United States of America,2012,109:11872-11877.
[3] HAREL-BEJI R,TZURI G,PORTNOY V ,et al.A genetic map of melon highly enriched with fruit quality QTLs and EST markers,including sugar and carotenoid metabolism genes[J].Theoretical and Applied Genetics,2010,121(3):511-533.
[4] 王贤磊,高兴旺,李冠,等.甜瓜遗传图谱的构建及果实与种子QTL分析[J].遗传,2011,33(12):1398-1408.
[5] 吕丽华.甜瓜果肉β-胡萝卜素含量的遗传分析及QTL定位[D]. 北京:中国农業科学院,2012.
[6] 杨光华,范荣,杨小锋,等.甜瓜果实颜色3个质量性状基因的定位[J].园艺学报,2014,41(5):898-906.
[7] D?AZ AUROR, BELKACEM Z,MOHAMAED F,et al.Mapping and introgression of QTL involved in fruit shape transgressive segregation into ‘Piel de Sapo melon (Cucucumis melo L.)[J]. PLoS ONE,2014,9(8):e104188.
[8] 高兴旺,王贤磊,宁雪飞,等.甜瓜掌状裂叶基因pll的精细定位[J].北方园艺,2015(6):98-102.
[9] 齐振宇.甜瓜株型和抗白粉病性状的遗传与全基因组关联分析[D].杭州:浙江大学,2015.
[10] 矫士琦.甜瓜果实、种子相关性状QTL分析[D].哈尔滨:东北农业大学,2017.
[11] 段金凤.甜瓜果实外观特征遗传分析及果皮颜色相关基因的初步定位[D].武汉:华中农业大学,2016.
[12] WANG Y H,WU D H,HUANG J H ,et al.Mapping quantitative trait loci for fruit traits and powdery mildew resistance in melon (Cucumis melo)[J] .Botanical Studies,2016,57:19.
[13] ARGYRIS J M,D?AZ A,VALENTINO R,et al.QTL Analyses in multiple populations employed for the fine mapping and identification of candidate genes at a locus affecting sugar accumulation in melon (Cucumis melo L.)[J].Frontiers in Plant Science,2017,8:1679.
[14] PEREIRA L,RUGGIERI V,P?REZ S ,et al.QTL mapping of melon fruit quality traits using a high-density GBS-based genetic map[J].BMC Plant Biology,2018,18:324.
[15] PALOMARES-RIUS F J,YUSTE-LIBONA F J,VIRUEL M A,et al.Inheritance and QTL mapping of glandular trichomes type idensity in Cucumis melo L.[J].Molecular Breeding,2016,36(9):132.
[16] ZHU H,SUN X,ZHANG Q,et al.GLABROUS (CmGL) encodes a HD-ZIP IV transcription factor playing roles in multicellular trichome initiation in melon[J].Theoretical and Applied Genetics,2017,131(2):1-11.
[17] 王学征,邱果,陈克农,等.基于BSA法开发CAPS标记定位甜瓜果面沟相关基因研究[J].东北农业大学学报,2018,49(5):17-23.
[18] CORDER E H,SAUNDERS A M,RISCH N J,et al.Protective effect of apolipoprotein E type 2 allele for late onset alzheimer disease[J].Nature Genetics,1994,7(2):180-184.
[19] 唐富福,徐非非,包劲松.全基因组关联分析在水稻遗传育种中的应用[J].核农学报,2012,27(5):598-606.
[20] TIAN F,BRADBURY P J,BROWN P J,et al.Genome-wide association study of leaf architecture in the maize nested association mapping population[J].Nature Genetics,2011,43(2):159-162.
[21] 马双武,刘君璞.甜瓜种质资源描述规范和数据标准[M].北京:中国农业出版社,2006.
[22] MCKENNA A,HANNA M,BANKS E,et al.The genome analysis toolkit: a mapreduce framework for analyzing next-generation DNA sequencing data[J].Genome Research,2010,20(9):1297-303.
[23] PRICE A L,PATTERSON N J,PLENGE R M,et al.Principal components analysis corrects for stratification in genome-wide association studies[J].Nature Genetics,2006,38(8):904-909.
[24] YU J,BUCKLER E S.Genetic association mapping and genome organization of maize[J].Current Opinion in Biotechnology,2006,17(2):155-160.
[25] CAO K,ZHOU Z,WANG Q,et al. Genome-wide association study of 12 agronomic traits in peach[J].Nature Communications,2016,7:13246.
[26] NIMMAKAYALA P,TOMASON Y R,ABBURI V L,et al.Genome-wide differentiation of various melon horticultural groups for use in gwas for fruit firmness and construction of a high resolution genetic map[J].Frontiers in Plant Science,2016,7:1437.
[27] 王艳玲,许彦宾,李琼,等.基于GWAS发掘甜瓜核心种质果实性状的关联位点[J].农业生物技术学报,2017,25(9):1434-1442.
- 探究企业会计核算的规范化管理方案
- 基层医疗卫生机构会计监督制度探讨构架
- 新政府会计制度对事业单位会计核算的影响分析
- 浅谈财务共享服务下管理会计的发展
- 关于企业合并报表会计问题研究
- 大数据背景下财务会计向管理会计转型研究
- 印刷企业成本管理问题及建议
- 新形势下企业会计信息化中存在的问题及对策研究
- 注册会计师职业服务市场的细分研究
- 解析政府会计准则和政府会计制度的特点及意义
- 新医院会计制度实施中的问题与对策探索
- 新医改形势下医院医保会计核算分析
- 对会计人员管理体制改革的原则与目标模式分析
- 对新企业会计准则下衍生金融工具会计处理的探讨
- 政府会计准则与高等学校会计制度的差异与对策思考
- 高速公路成本分析及控制策略
- 会计集中核算实际操作中的变化及风险防范
- 新政府会计制度对政府会计核算工作的影响及建议
- 浅析县级公立医院成本核算存在的问题及对策
- 广播电视行业成本核算管理问题及措施探究
- 公立医院执行政府会计制度改革的衔接工作分析
- 从内控角度看房地产公司成本控制
- 浅析房地产开发公司成本管理存在问题与解决方案
- 电子制造业目标成本管理研究
- 浅议新形势下代理记账行业的发展与创新
- baboons'
- baboons
- baby
- baby boom
- babyboom
- babyboomer
- baby-boomer
- baby boomer
- baby boomer
- baby boomers
- baby booms
- baby-buggy
- babycarriage
- baby carriage
- baby carriages
- babyhood
- babyhoods
- babying
- babyish
- babyishly
- babyishness
- babyishnesses
- babyishnesses'
- babyishness's
- babyism
- 将文字中的某些部分删除
- 将旗
- 将无
- 将无作有
- 将无做有
- 将昃
- 将明之义
- 将明之材
- 将星拱北辰
- 将星陨
- 将是兵中胆
- 将是兵之胆,兵是将之威
- 将晚
- 将朝赠言
- 将本图利
- 将本求利
- 将机就机
- 将机就计
- 将权力交给别人
- 将李代桃
- 将来
- 将来不会再有见面的机会
- 将来佳器
- 将来很有发展前途的人才
- 将来有那么一天:一旦 有一天 有朝一日(现在以后的时间