刘伟莹 张雨晴 王丹玉 李佳妮 杨成君

摘要:为了探索MADS基因家族的耐盐胁迫功能,从以吉尔吉斯白桦(Betula kirghisorum)23号为试验材料测得的转录组文库中筛选出28个吉尔吉斯白桦的MADS-box基因序列对其编码蛋白的基本理化性质、亚细胞定位、二级结构、跨膜结构和信号肽、基因结构、保守基序、蛋白系统进化等方面进行初步生物信息学分析,并在NaHCO3胁迫下对其表达情况进行分析。结果显示,除蛋白BkMADS1和BkMADS20外,其余均为亲水性蛋白;在28个吉尔吉斯白桦MADS蛋白中有19个定位在细胞核中;蛋白BkMADS7和BkMADS12的二级结构组成以无规则卷曲为主,其余蛋白则以α-螺旋为主;所有蛋白均没有信号肽,即全为非分泌性蛋白。此外,进化分析结果表明,MADS盒为MADS-box转录因子高度保守的结构域,大多数吉尔吉斯白桦MADS蛋白为Ⅱ型。在0.6% NaHCO3胁迫下,15个BkMADS基因上调表达,13个BkMADS个基因下调表达。
关键词:吉尔吉斯白桦;MADS基因家族;MADS-box转录因子;转录组文库;非分泌性蛋白;生物信息学;NaHNO3胁迫;基因表达
中图分类号:Q943.2;R857.3;S792.153.01 ?文献标志码: A ?文章编号:1002-1302(2020)10-0085-09
收稿日期:2019-04-27
基金项目:东北林业大学大学生创新训练项目(编号:201810225478)。
作者简介:刘伟莹(1999—),女,黑龙江巴彦人,主要从事森林植物资源学研究研究。E-mail:247335605@qq.com。
通信作者:杨成君,硕士,副教授,主要从事森林植物资源学研究。E-mail:nxyycj@sina.cn。
MADS基因家族是目前研究最广泛的植物转录因子之一,具有多种生物功能,广泛参与调控植物生长发育的多个过程和逆境应答。1990年,MADS-box 转录因子首先在金鱼草中被发现[1],它是一类序列特异的转录调控因子,主要在激活或抑制基因的转录反应过程中起作用[2]。MADS-box基因编码的蛋白即为MADS-box转录因子,是MADS基因功能的直接行使者,通常以同源或异源二聚体的形式使其保守结构域与特定的DNA序列结合,从而调控基因的表达[3-4]。MADS-box转录因子主要由MADS盒、K盒、I区、C末端、N末端5个部分组成。MADS盒是由约57个氨基酸组成的高度保守结构域,主要与DNA结合,有时也形成二聚体或与辅助因子结合[4-7];K盒由约70个氨基酸组成,是MADS-box转录因子的特征序列,也是发生二聚体化的结构单元;I区位于MADS盒和K盒之间,是由31~35个氨基酸组成的非保守区域,该区域内含有较多的亲水残基,它们的作用是帮助二聚体化的转录因子与DNA结合形成复合体;C末端位于K盒的下游,是由约30个氨基酸组成的非保守区域,该区域内富含疏水残基;N末端位于MADS盒的上游,是富含碱性氨基酸的亲水结构域[4,8]。
MADS由4种蛋白因子基因[MCM1)(mini chromosome maintenance 1)、AGAMOUS、DEFICIENS和SRF(serum response factor]的首写字母构成[8]。其中,MCM1是酿酒酵母(Saccharomyces cerevisiae)特异性基因的转录控制中心,可以参与调节细胞周期和细胞的生长代谢过程;AGAMOUS(AG)和DEFICIENS(DEF)分别是拟南芥(Arabidopsis thaliana)和金鱼草(Antirrhinum majus)花器官特征基因的产物;SRF是人类(Homo sapiens)的血清应答因子,涉及原癌基因的协同转录[3,5]。这4种蛋白因子基因的共同特点是都有1个MADS盒[9]。MADS-box基因家族中所有的基因都拥有一个长度约为180 bp、能够编码MADS-box结构域的高度保守核苷酸序列,因此被称为MADS-box基因[10]。
MADS-box基因广泛存在于动物、植物和真菌中,其序列十分保守,因此功能特异性强。MADS-box蛋白在真核生物的生长发育过程中发挥着重要作用,特别是在显花植物的花器官分化、开花时间的调节以及相关的果实发育与成熟等方面[11-13]。MADS-box蛋白在植物花发育过程中的调控功能根据器官决定因子可以归纳为花器官发育的ABCDE模型,其中A、E功能基因调控萼片的发育,A、B、E功能基因调控花瓣的发育,B、C、E功能基因调控雄蕊的发育,C、E功能基因调控心皮的发育,C、D、E功能基因调控胚珠的发育,A、C功能基因則相互拮抗[14-16]。在果实成熟过程的调控方面,陈翠翠等在番茄中发现了2个与果实成熟过程密切相关的MADS-box基因,并将其分别命名为LeMADS-RIN和LeMADS-MC,前者调控果实的成熟过程,而后者则在萼片的发育和花序的决定方面发挥作用[2,17]。此外,研究表明,拟南芥在受精后的裂片分化过程中需要FUL基因,该基因的过量表达可抑制裂片区域的木质化,且该基因负调控SHP基因,如果它发生突变,则会导致果实不能正常开裂[2,18]。MADS-box基因的特异性作用与植物所处的内外环境因素密切相关[19]。NO-3作为信号分子,可以刺激拟南芥侧根的伸长生长,该刺激作用主要依赖于MADS基因家族中ANR1基因的表达[19-20]。MADS-box基因不仅在分化期的花器官原基中表达,在植物的其他部位也有表达[21],可调控根的生长以及根瘤的形成、调节叶序的发生和转变、影响春化、调节顶端分生组织的分化、调节胚珠的发育、调控光合作用和营养代谢等多个过程[22]。
1 材料与方法
1.1 试验材料
从以吉尔吉斯白桦(Betula kirghisorum)23号为试验材料测得的转录组文库中筛选出吉尔吉斯白桦的MADS-box基因序列,利用在线软件ORF Finder(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)查找出相应的编码序列,并将其翻译为氨基酸序列。
1.2 试验方法
1.2.1 吉尔吉斯白桦MADS蛋白的理化性质和亚细胞定位分析 利用在线软件ProtParam tool(http://web.expasy.org/prot param/)测定MADS蛋白的理化参数(氨基酸数目、分子量、理论等电点、脂肪族氨基酸指数、疏水性)。通过软件The WoLF PSORT(http://www.genscript.com/psort/wolf_psort.html)对MADS蛋白进行亚细胞定位。
1.2.2 吉尔吉斯白桦MADS蛋白的二级结构预测 运用常用的生物信息学在线软件SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)预测MADS蛋白的二级结构。
1.2.3 吉尔吉斯白桦MADS蛋白跨膜结构域和信号肽的预测 采用在线软件TMPred(http://www.ch.embnet.org/software/TMPRED_form.html)对吉尔吉斯白桦的MADS蛋白进行跨膜结构域预测;采用在线软件SignalP 4.1(http://www.cbs.dtu.dk/ services/SignalP/)预测MADS蛋白中是否含有信号肽。
1.2.4 吉尔吉斯白桦MADS基因结构的预测 根据所得到的吉尔吉斯白桦MADS基因序列和编码序列,用基因结构显示服务器GSDS2.0(http://gsds.cbi.pku.edu.cn/)分析MADS基因的结构。
1.2.5 吉尔吉斯白桦MADS蛋白的多重序列对比和保守基序(motif)识别 运用Clustalx v1.83软件对吉尔吉斯白桦的MADS蛋白进行多重序列对比分析。利用在线软件MEME(http://meme-suite.org/tools/meme)分析吉尔吉斯白桦MADS蛋白的氨基酸序列保守基序,参数设置如下:同一基序在1条系列中出现0次或1次,基序最大发现值为5,长度范围为70个氨基酸残基,其他参数为默认值。
1.2.6 吉尔吉斯白桦MADS蛋白系统的进化分析 本试验结合拟南芥对吉尔吉斯白桦的MADS蛋白系统进行进化分析。从JGI Phytozome v11.0(https://phytozome.jgi.doe.gov/pz/portal.html)数据库中下载得到拟南芥MADS蛋白的氨基酸序列,利用MEGA 5.1软件中的邻位相接法(NJ)[自举值(bootstrap)(1 000次重复)]构建吉尔吉斯白桦和拟南芥MADS蛋白的系统进化树,进而对吉尔吉斯白桦的MADS蛋白系统进行进化分析和分类。
1.2.7 NaHCO3胁迫下吉尔吉斯白桦MADS基因家族的表达分析 以采用0.6% NaHCO3溶液盐胁迫48 h的吉尔吉斯白桦叶片为试验材料,经检测计算得到log2(RPKMBk/RPKMCK)值[RPKM表示每100百万读长(reads)中来自于某基因每1 000碱基长度的读长数;Bk是吉尔吉斯白桦拉丁文的缩写,CK是对照],通过柱形图对盐胁迫下吉尔吉斯白桦叶片中MADS基因家族的表达进行分析。
2 结果与分析
2.1 吉尔吉斯白桦MADS蛋白的理化性质和亚细胞定位分析
本试验从吉尔吉斯白桦23号的转录组文库中共得到28个MADS-box基因序列,从表1可以看出,这28个基因的序列长度和所编码的氨基酸数目均存在着较大的差异。氨基酸数目在38~352个范围内;BkMADS12的氨基酸数目和分子量最大,分别为352个和39 899.1 u; BkMADS7的氨基酸数目最少,分子量最小,分别为38个和4 443.2 u。28个MADS蛋白的等电点变化范围较大, 在4~12之间均有分布,其中有10个蛋白的等电点在9~10之间;BkMADS4的等电点最高,为11.57;BkMADS1的等电点最低,为4.63。除BkMADS1和BkMADS2外,其他所有蛋白的脂肪族氨基酸指数都小于100,说明大多数吉尔吉斯白桦的MADS蛋白為亲水性蛋白。28个吉尔吉斯白桦MADS蛋白的疏水性平均值(GRAVY)预测结果有正有负,其中除BkMADS1和BkMADS20外,其余蛋白的疏水性平均值均为负。
本试验利用The WoLF PSORT软件对吉尔吉斯白桦的28个MADS蛋白进行亚细胞定位预测,预测结果以得分的形式表现出来,分值越高,表示预测结果的准确性越大。但该软件具有一定的局限性,只能预测以甲硫氨酸(蛋氨酸,M)开头的氨基酸序列,因此本试验中氨基酸序列以亮氨酸(L)开头的BkMADS1、BkMADS2、BkMADS7、BkMADS10、BkMADS18、BkMADS23、BkMADS24蛋白则无法用该软件进行亚细胞定位。从亚细胞定位的预测结果(表2)可以看出,BkMADS3、BkMADS4、BkMADS5、BkMADS6、BkMADS8、BkMADS9、BkMADS11、BkMADS12、BkMADS13、BkMADS14、BkMADS15、BkMADS16、BkMADS17、BkMADS19、BkMADS21、BkMADS22、BkMADS25、BkMADS27、BkMADS28在细胞核(nucleus)中的预测分值最大;BkMADS26在细胞质(cytoplasm)、细胞质与细胞核之间空间的预测分值相同;而BkMADS20在细胞质和细胞核之间空间位置的预测分值最大。
2.2 吉尔吉斯白桦MADS蛋白的二级结构预测
蛋白质的二级结构是指它的多肽链中有规则重复的构象,主要形式包括α-螺旋(α-helix)、β-折叠(β-sheet)、β-转角(β-turn)和无规则卷曲(random coil)。目前预测蛋白质二级结构的方法逐渐增多,其中基于对准的自优化预测(self optimized prediction method from alignment,SOPMA)方法的输出形式比较直观,因此本试验采用SOPMA方法对吉尔吉斯白桦MADS蛋白的二级结构进行预测。SOPMA采用5种方法对蛋白的二级结构进行预测,然后将结果进行优化组合,最后汇集整理成一个一致的预测结果。蛋白二级结构的预测采用四态定义,即α-螺旋、β-折叠、β-转角和无规则卷曲。吉尔吉斯白桦MADS蛋白的二级结构预测结果(表3)表明,在BkMADS7、BkMADS12蛋白的二级结构中所占比例最大的是无规则卷曲,而在其余蛋白的二级结构中α-螺旋所占比例总体最大;大多数蛋白的二级结构组成百分比表现为α-螺旋>无规则卷曲>β-折叠>β-转角,而在BkMADS3蛋白中没有出现β-折叠,在BkMADS13蛋白中α-螺旋和β-折叠所占比例相同,在BkMADS20蛋白中β-转角和无规则卷曲所占比例相同。
2.3 吉尔吉斯白桦MADS蛋白跨膜结构域和信号肽的预测
蛋白质典型的跨膜螺旋区域主要由20~30个疏水性氨基酸(如亮氨酸、异亮氨酸、缬氨酸、甲硫氨酸、甘氨酸、丙氨酸等)组成。从表4可以看出,在28个吉尔吉斯白桦MADS蛋白中有16个具有跨膜螺旋结构,而在这16个具有跨膜结构蛋白中有11个具有1个内部向外螺旋和1个外部向内螺旋结构;在BkMADS8、BkMADS9、BkMADS17、BkMADS22蛋白中仅仅具有1个内部向外螺旋结构,而不具有外部向内螺旋结构;在BkMADS10蛋白中具有2个内部向外螺旋和1个外部向内螺旋结构,是这28个蛋白中结构较特殊的一种蛋白。
信号肽(signal peptide)常常位于分泌蛋白的N端,一般由15~30个氨基酸构成,其中包含6~15个带正电荷的非极性氨基酸,可以用于判断一个蛋白质是否为分泌蛋白,且与蛋白质在细胞内的定位有关。通过软件SignalP ?4.1得出的信号肽预测结果表明,吉尔吉斯白桦的28个MADS基因编码的蛋白均没有信号肽序列,说明吉尔吉斯白桦的28个MADS蛋白均为非分泌性蛋白。
2.4 吉尔吉斯白桦MADS基因结构的预测
吉尔吉斯白桦MADS基因结构的预测结果如图1所示,由于本试验没有得到吉尔吉斯白桦28个MDAS基因的基因组数据,只有其转录组数据,因此在预测基因结构时只能预测到外显子的位置,即非翻译区位置,无法显示出内含子的情况。
2.5 吉尔吉斯白桦MADS蛋白的多重序列对比和保守基序识别
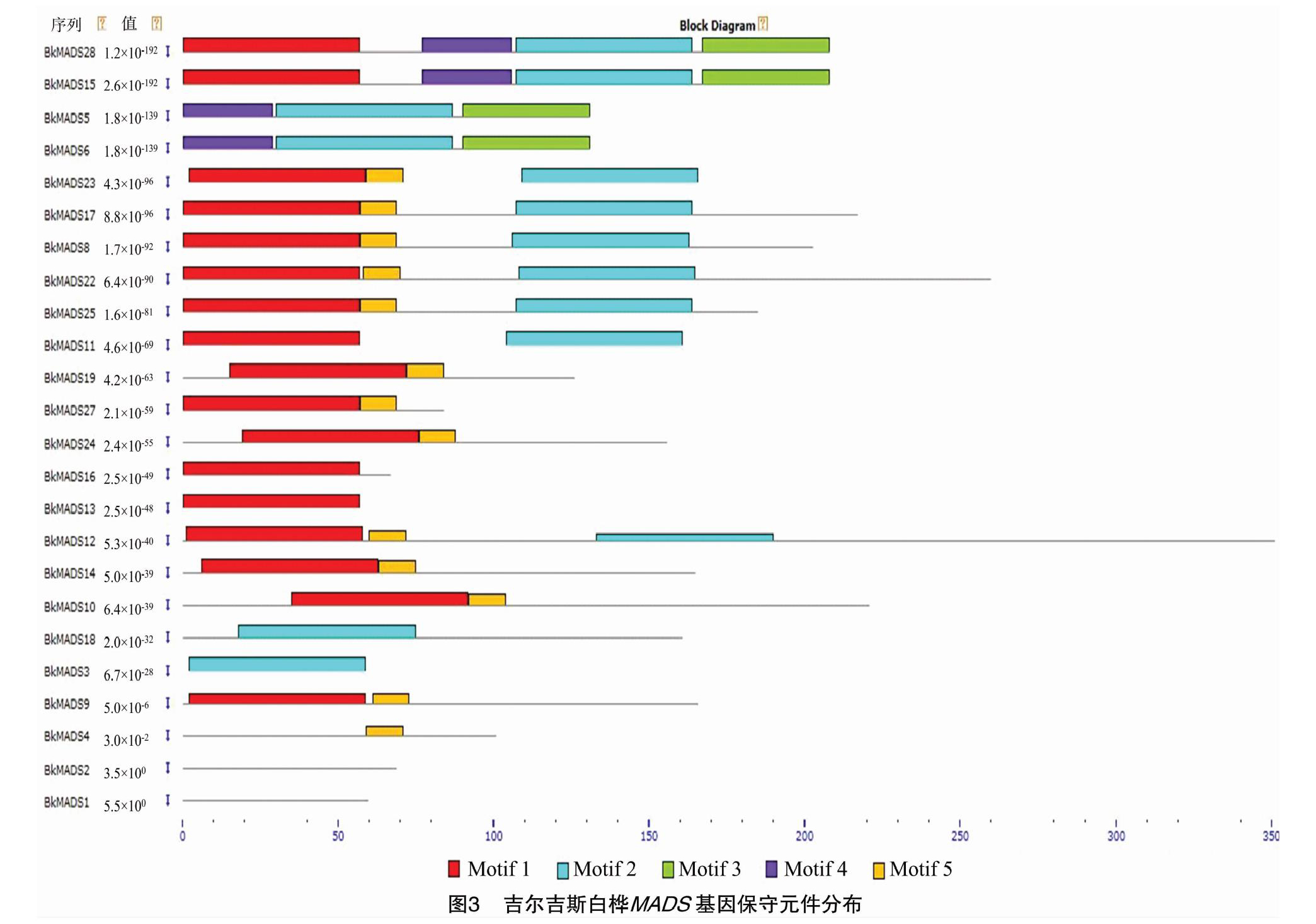
利用Clustalx v1.83软件对吉尔吉斯白桦MADS蛋白的氨基酸序列进行多重对比分析,结果(图2)表明, 28个吉尔吉斯白桦MADS基因家族基
因的MADS-box结构域高度保守。利用MEME在线软件分析28个吉尔吉斯白桦MADS蛋白的氨基酸保守基序,结果(图3)表明,MADS蛋白包含5个保守元件, 其中基序1含有57个氨基酸,且大多数吉尔吉斯白桦MADS蛋白的氨基酸序列中均含有该基序,所以推测其是转录因子MADS-box的典型结构MADS盒;在MADS盒之后的是Ⅰ区,包含基序5,最佳匹配序列是SSSSMQKVIERY;Ⅰ区之后是K区,包含基序2;而BkMADS5、BkMADS6、BkMADS15、BkMADS28蛋白所含有的基序3、4则为未知基序。本试验中,由于得到的BkMADS1、BkMADS2、BkMADS7、BkMADS20、BkMADS21、BkMADS26基因序列长度较短,未能识别出其保守氨基酸的基元序列;图3显示,基因BkMADS3、BkMADS4、BkMADS5、BkMADS6、BkMADS18中不含有保守结构域MADS盒, 可能不属于MADS基因家族,同时由于本试验缺乏基因组数据,无法准确地判断其分类,因此在以下分析中不再对基因BkMADS1、BkMADS2、BkMADS3、BkMADS4、BkMADS5、BkMADS6、BkMADS7、BkMADS18、BkMADS20、BkMADS21、BkMADS26进行研究。
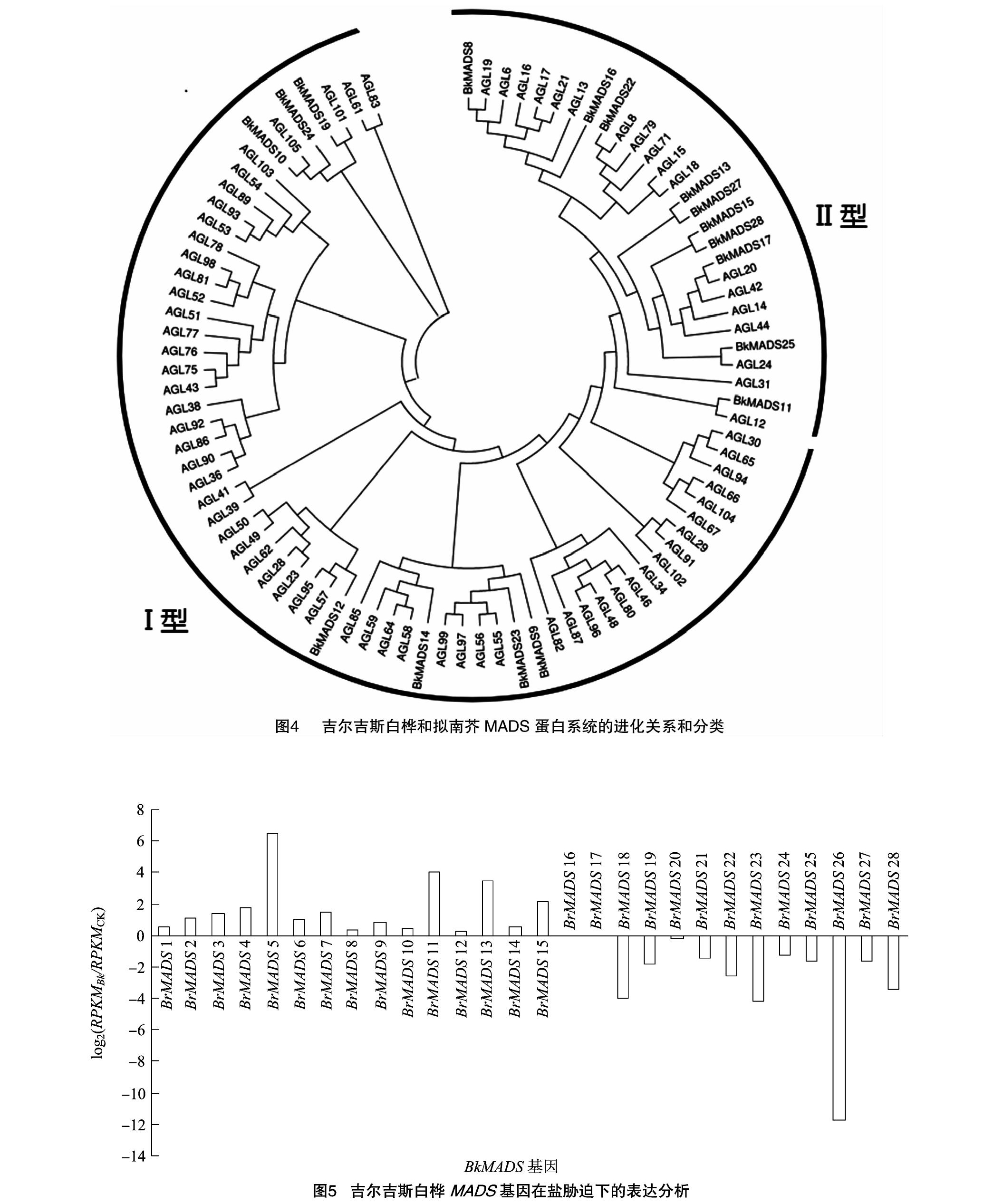
2.6 吉尔吉斯白桦MADS蛋白系统的进化分析
利用进化树分析软件MEGA5.1对吉尔吉斯白桦MADS蛋白和74个拟南芥AGL型MADS蛋白进行系统进化树构建,并依据Parenicová等對拟南芥AGL型MADS蛋白的分类[23]进行进化关系分析和分类。结果(图4)显示,在所研究的17个吉尔吉斯白桦MADS蛋白中有10个(BkMADS8、BkMADS11、BkMADS13、BkMADS15、BkMADS16、BkMADS17、BkMADS22、BkMADS25、BkMADS27、BkMADS28)属于Ⅱ型,7个(BkMADS9、BkMADS10、BkMADS12、BkMADS14、BkMADS19、BkMADS23、BkMADS24)属于Ⅰ型。由于本试验只有转录组数据,缺少基因组数据,因此无法进一步地对Ⅰ型中的MADS蛋白进行分类。
2.7 NaHCO3胁迫下吉尔吉斯白桦MADS基因家族的表达分析
从图5可以看出,在盐胁迫下,吉尔吉斯白桦叶片中有15个BkMADS基因上调表达,而13个BkMADS基因的表达受到抑制,呈现下调表达趋势,其中BkMADS5、BkMADS11、BkMADS13的上调表达趋势明显,BkMADS26、BkMADS18、BkMADS23、BkMADS28的表达被明显抑制。在盐胁迫下,各BkMADS基因的表达情况存在明显差异,因此须要进一步研究它们的表达特性。
3 结论
本研究通过分析吉尔吉斯白桦叶片中MADS基因家族的转录组数据获得28个具有完整ORF的MADS-box转录因子,并利用转录组数据分析其所编码蛋白的理化性质、亚细胞定位、二级结构、跨膜结构和信号肽、基因结构、保守结构域特征、系统进化分类以及在盐胁迫下的表达特性。结果表明,28个吉尔吉斯白桦MADS-box基因所编码的氨基酸数目在30~360个范围内;等电点在4~12之间均有分布;大多数MADS-box蛋白为亲水性蛋白,亚细胞定位在细胞核中,且α-螺旋在二级结构的预测结果中所占比例最大;所有的MADS-box蛋白均为非分泌性蛋白;采用MEME软件对氨基酸保守基序进行识别分析,得到17个有意义的MADS-box基因,主要可以分为两大类,其中大多数属于Ⅱ型;在盐胁迫下,15个BkMADS基因上调表达,13个BkMADS基因下调表达。表明在吉尔吉斯白桦的MADS基因家族中一部分基因具有一定的耐盐性。
参考文献:
[1]马忠强. MADS转录因子对植物发育的重要作用[J]. 种子世界,2014(5):24-26.
[2]陳翠翠,马元武,冯永君,等. MADS-box家族蛋白在植物开花、结实及根瘤形成中的多功能调节作用[J]. 华北农学报,2008,23(增刊2):74-77.
[3]侯传明. 山核桃MADS转录因子的H2Y分析和活性组织表达水平检测[D]. 杭州:浙江农林大学,2014.
[4]胡丽芳,金志强,徐碧玉. MADS-box基因对花的发育及开花早晚的影响[J]. 生命科学研究,2004,8(4):7-12.
[5]Shore P,Sharrocks A D. The MADS-box family of transcription factors[J]. European Journal Biochemistry,1995,229(1):1-13.
[6]Krizek B A,Meyerowitz E M. Mapping the protein regions responsible for the functional specificities of the Arabidopsis MADS domain organ-identity proteins[J]. Proceeding of the National Acad Sciences,1996,93(9):4063-4070.
[7]Jack T,Brockman L L,Meyerowitz E M. The homeotic gene APETALA 3 of Arabidopisis thaliana encodes a MADS-box and is expressed in petals and stamens[J]. Cell,1992,68(4):683-697.
[8]Schwarz-Sommer Z,Huijser P,Nacken W,et al. Genetic control of flower development by homeotic genes in Antirrhinum majus[J]. Science,1990,250(4983):931-936.
[9]Zachgo S,Saedler H,Schwarz-Sommer Z. Pollen-specific expression of DEFH125,a MADS-box transcription factor in Antirrhnum with unusual features[J]. The Plant Journal,1997,11(5):1043-1050.
[10]崔荣峰,孟 征. 花同源异型MADS-box基因在被子植物中的功能保守性和多样性[J]. 植物学通报,2007,24(1):31-41.
[11]Messenguy F,Dubois E. Role of MADS-box proteins and their cofactors in combinatorial control of gene expression and cell development[J]. Gene,2003,316:1-21.
[12]Irish V F. The evolution of floral homeotic gene function[J]. Bioessays,2003,25(7):637-645.
[13]李元元,王 鲁,苏振刚,等. MADS-box基因控制植物成花的分子机理[J]. 基因组学与应用生物学,2010,29(6):1122-1132.
[14]Kaufmann K,Melzer R,Theissen G. MIKC-type MADS-domain proteins:structural modularity,protein interactions and network evolution in land plants[J]. Gene,2005,347(2):183-198.
[15]Theissen G,Saedler H. Floral quartets[J]. Nature,2001,409:469-471.
[16]徐启江,关录飞,吴笑女,等. 草原龙胆MADS-box基因的克隆及表达分析[J]. 植物学通报,2008,25(4):415-429.
[17]Vrebalov J,Ruezinsky D,Padmanabhan V,et al. A MADS-box gene necessary for fruit ripening at the tomato ripening-inhibitor (rin) locus[J]. Science,2002,296(5566):343-346.
[18]胡丽芳,金志强,徐碧玉. MADS-box基因在果实发育、成熟过程中的作用[J]. 分子植物育种,2005,3(3):415-420.
[19]马 辉,张智俊,罗淑萍. 植物MADS-box基因研究进展[J]. 生物技术通报,2006(6):14-18.
[20]Zhang H M,Brian G,Forde. An Arabidopsis MADS-box gene that controls nutrient-induced changes in root architecture[J]. Science,1998,279(16):407-409.
[21]李 娟,薛庆中. 拟南芥及水稻转录因子MADS密码子的偏好性比较[J]. 浙江大学学报(农业与生命科学版),2005,31(5):513-517.
[22]刘菊华,徐碧玉,张 静,等. MADS-box转录因子的相互作用及对果实发育和成熟的调控[J]. 遗传,2010,32(9):893-902.
[23]Parenicová L,de Folter S,Kieffer M,et al. Molecular and phylogenetic analyses of the complete MADS-box transcription factor family in Arabidopsis:new openings to the MADS world[J]. The Plant Cell,2003,15(7):1538-1551.
- 浅谈小型化微波铁氧体隔离器的制作
- IL10基因rs1800871位点多态性与结直肠癌发病风险的关系
- BIM技术在南京某大楼幕墙中的应用
- 基于农作物安全情况下无公害栽培的管理技术分析
- 基于词频分析的新农人创业政策研究
- 交通运输维修行业的管理对策
- 浅析农民职业教育中英语能力的培养
- 新媒体技术下科技馆科学教育的创新转变
- 离退休干部思想政治工作中的人文关怀和心理疏导研究
- 京津冀一体化秦皇岛市导游队伍培训问题研究
- 儒家角色伦理学视域下的师生关系
- 历史教学中践行核心素养“家国情怀”
- 微课在高中生物教学中的应用
- 基于语境分析的词汇教学方法探析
- 将中学生的学习小聪明转化为大智慧
- “教学相长”师生学习共同体建设的校本实践
- 问渠把脉 活水自来
- 注重培养学生思维能力
- 班主任应用爱心为学生撑起一片天空
- 时事政治在初中政治课堂教学中的应用探析
- 提高课堂教学效率,从“头”开始
- 我终于成长了
- 初一学生班级心理健康教育课程研究
- 浅谈物理课堂中转变学生前概念的教学策略
- 酸碱盐的复习案例分析
- decibels
- decidabilities
- decidability
- decide
- decided
- decidedly
- decidedness
- decidednesses
- decidedness's
- decide on
- decide on sth
- deciders
- decides
- deciding
- deciduous
- deciduously
- deciduousness
- deciduousnesses
- decimal
- decimal point
- decimal points
- decimals
- decimal²
- decimal¹
- decimate
- 尚父鹰扬
- 尚犹
- 尚玄
- 尚甫威虞,苍兕明誓
- 尚祈善摄
- 尚祈善摄,勿过悲戚
- 尚祈时赐教言
- 尚祈节哀
- 尚祈节哀,以慰泉壤
- 尚祈鉴其愚忱,是所深感
- 尚章
- 尚纲斋集
- 尚羊
- 尚考
- 尚自珍摄
- 尚节
- 尚荷鼎力
- 尚蒙慨诺,不胜感激
- 尚虚中馈
- 尚请保重
- 尚请节哀
- 尚贤使能
- 尚贤者,政之本也
- 尚还
- 尚飨