糖蛋白糖基化位点及糖型的多重质谱分析
于晶等
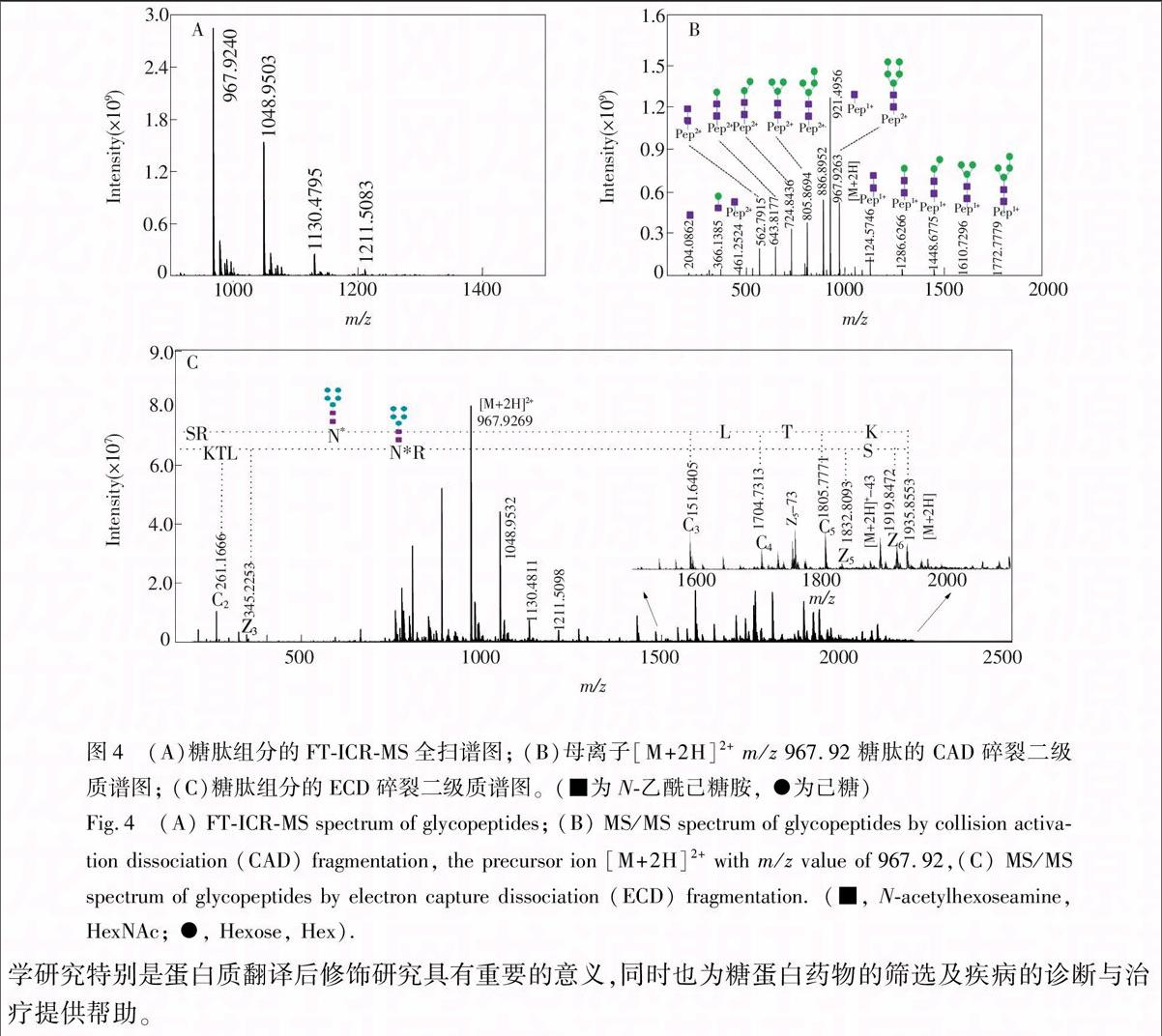


摘 要 以糖蛋白核糖核酸酶B(RNase B)为研究对象,采用基质辅助激光解吸电离串联飞行时间质谱(MALDITOF/TOFMS)、傅里叶变换离子回旋共振质谱(FTICRMS)的碰撞活化解离(CAD)和电子转移解离(ECD)多种质谱技术对RNase B的糖肽的完整信息进行解析。通过多种质谱相互验证,确定了RNase B的糖肽组分的氨基酸序列为SRNLTK、糖链结构为(HexNAc)2(Hex)5(HexNAc)2(Hex)9及糖基化位点为第34位氨基酸残基。本方法为糖蛋白结构的全面解析提供了一种快速、有效、全面的研究思路。
关键词 基质辅助激光解吸电离串联飞行时间质谱; 傅里叶变换离子回旋共振质谱; 碰撞活化解离; 电子捕获解离; 核糖核酸酶B
1 引 言
蛋白质的糖基化是糖链在糖基转移酶作用下结合到蛋白质特定氨基酸残基上[1],是蛋白质的一种重要的翻译后修饰方式[2]。目前, 已知哺乳动物蛋白质中至少有1/2是糖蛋白,广泛分布于各种组织和细胞中[3]。糖蛋白糖链的结构多样,功能复杂,并且由于生物合成的非模板性和结构的微不均一性,使糖链具有了更多的分支结构、连接方式和空间构象,因此,修饰后的糖蛋白会产生多种生物活性,对生物体的生长、发育甚至对生物体的生存起着至关重要的作用[4]。蛋白质的糖基化类型主要为N连接和O连接的糖基化,N连接的糖基化发生在特定的氨基酸序列AsnXSer/Thr,其中X是除脯氨酸以外的任意氨基酸。O连接的糖基化发生在Ser/Thr氨基酸序列上。蛋白质糖基化与许多疾病的发展密切相关,在某些疾病状态下,聚糖的相对丰度和分支结构与正常相比发生了变化[5],这些异常的变化可为疾病的早期诊断及治疗提供帮助[6]。
常规的糖蛋白研究方法主要有基质辅助激光解吸电离飞行时间质谱(MALDITOFMS)[7]、电喷雾质谱(ESIMS)[8]、傅里叶变换离子回旋共振质谱(FTICRMS)[9]等。MALDITOFMS和ESIMS这两种方法都是对糖链和肽段分别研究,以得到糖基化位点和糖链结构信息,或者是在完整糖肽水平上对糖蛋白结构进行解析。但是在对完整糖肽进行解析时,得到的主要是糖链的结构信息,糖肽序列信息较少,导致位点鉴定困难,尤其是对于未知的样品。完整糖蛋白的全面解析对于疾病的诊断与治疗、糖蛋白药物的筛选及蛋白质组学研究具有重要意义。因此,开发完整糖肽水平的研究方法显得尤为重要。FTICRMS的碰撞诱导解离(CAD)可以得到糖链的结构信息,电子捕获解离(ECD)碎裂方式通过捕获一个自由电子,使其与质子化的多电荷肽段相互作用,从而诱发多肽主链在NCα键处发生断裂,主要产生C、Z离子,在碎片离子上保留修饰基团为翻译后修饰位点的鉴定提供了重要的信息[10,11]。MALDITOF/TOFMS样品需求量少,且提供丰富的低分子段碎片离子信息。因此,采用多重质谱的联合分析为完整糖肽的全面研究提供可能。
本研究以糖蛋白核糖核酸酶B(RNase B)的完整糖肽为研究对象,采用液相色谱分离富集,联合MALDITOF/TOFMS和FTICRMS的CAD和ECD解离模式等多种质谱技术,在解析糖链结构的同时,获得肽段序列和糖基化位点的信息,实现糖蛋白结构的全面、同步解析,为未知及复杂糖蛋白的完整糖肽的解析提供参考方法。
4 结 论
本研究采用液相色谱对样品中的糖肽进行分离富集,MALDITOF/TOFMS和FTICRMS多重质谱检测,对糖蛋白糖链结构及糖基化位点进行同步解析。MALDITOF/TOFMS通过对少量样品的完整糖肽及切除糖链的肽骨架分析,可获得肽骨架序列、糖基化位点及糖链结构的信息,分析速度快。FTICRMS的CAD碎裂模式和ECD碎裂模式相结合,不需要切除糖链即可实现肽段序列、糖基化位点及糖链结构的同时分析,如与nanoLC联用,可望进行糖肽的定量分析。多种质谱技术联合及相互验证,鉴定RNase B的糖链结构为(HexNAc)2(Hex)5(HexNAc)2(Hex)9,糖基化位点为第34位氨基酸残基,糖肽氨基酸序列为SRNLTK。本研究为复杂糖蛋白的结构解析提供了一种简单、有效的方法,对于蛋白质组学研究特别是蛋白质翻译后修饰研究具有重要的意义,同时也为糖蛋白药物的筛选及疾病的诊断与治疗提供帮助。
References
1 WANG Sheng, ZOU Xia, ZHANG Yan. Progr Chem., 2010, 22(12): 2428-2435
王 胜, 邹 霞, 张 延. 化学进展, 2010, 22(12): 2428-2435
2 SHI YuMei, ZHENG Lei, LI Juan. Progr Mod。 Biomed., 2011, 11(24): 5190-5192
石玉梅, 郑 磊, 李 娟. 现代生物医学进展, 2011, 11(24): 5190-5192
3 WANG LinJie, ZHENG DeXian, GAO Youhe. Basic Clin. Med., 2007, 27(2): 122-128
王林杰, 郑德先, 高友鹤. 基础医学与临床, 2007, 27(2): 122-128
4 XU Sha, ZHANG Ping, HUANG LinJuan, WANG ZhongFu. Chem. J. Chinese Universities, 2010, (31)10: 1992-1998
徐 莎, 张 萍, 黄琳娟, 王仲孚. 高等学校化学学报, 2010, (31)10: 1992-1998
5 CHEN Gang, BAI Quan, GENG XinDu. Chinese Journal of Chromatography, 2006, 24(5): 425-431
陈 刚, 白 泉, 耿信笃. 色谱, 2006, 24(5): 425-431
6 Alley W R, Jr, Mechref Y, Novotny M V. Rapid Commun. Mass Spectrom., 2009, 23(1): 161-170
7 Harvey D J, Royle L, Radcliffe C M, Rudd P M, Dwek R A. Anal. Biochem., 2008, 376(1): 44-60
8 Nwosu C C, Seipert R R, Strum J S, Hua S S, An H J, Zivkovic A M, German B J, Lebrilla C B. J. Proteome Res., 2011, 10(5): 2612-2624
9 Hongo Y, Nakamura T, Sato A. J. Mass Spectrom. Soc. Jpn., 2007, 55(2): 77-82
10 SUN RuiXiang, DONG MengQiu, CHI Hao, YANG Bing, XIU LiYun, WANG LeHeng, FU Yan, HE SiMin. Progr. Biochem Biophys., 2010, 37(1): 94-102
孙瑞祥, 董梦秋, 迟 浩, 杨 兵, 秀丽蕴, 王乐珩, 付 岩, 贺思敏. 生物化学与生物物理进展, 2010, 37(1): 94-102
11 LIU GuangXian, GUAN ShanHong, WANG Hui, FENG JianXiong. Food Sci., 2013, 34(17): 43-46
刘光宪, 管珊红, 王 辉, 冯健雄. 食品科学, 2013, 34(17): 43-46
12 ZENG Rong, SHAO XiaoXia, WANG KeYi, XIA QiChang. Acta Biochem. Bioph. Sinica, 1999, 31(6): 695700
曾 嵘, 邵晓霞, 王克夷, 夏其昌. 生物化学与生物物理学报, 1999, 31(6): 695-700
13 Zhao P, Viner R, Teo C F, Boons GJ, Horn D, Wells L. J. Proteome Res., 2011, 10(9): 4088-4104
14 ZHOU Wei, LIU XiaoHui, ZHOU XinWen, SHEN HuaLi, YANG PengYuan. Chinese Journal of Chromatography, 2007, 25(5): 623-627
周 玮, 刘晓慧, 周新文, 申华莉, 杨芃原. 色谱, 2007, 25(5): 623-627
15 CHEN YaoHan, YAN GuoQuan, ZHOU XinWen, YANG PengYuan. Chinese Journal of Chromatography, 2010, 28(2): 135-139
陈瑶函, 晏国全, 周新文, 杨芃原. 色谱, 2010, 28(2): 135-139
16 An H J, Peavy T R, Hedrick J L, Lebrilla C B. Anal. Chem., 2003, 75(20): 5628-5637
17 Singh C, Zampronio C G, Greese A J, Cooper H J. J. Proteome Res., 2012, 11(9): 4517-4525
18 JIANG Jing, YING WanTao, QIAN XiaoHong. Chinese J. Anal. Chem., 2014, 42(2): 159-165
江 静, 应万涛, 钱小红. 分析化学, 2014, 42(2): 159-165



摘 要 以糖蛋白核糖核酸酶B(RNase B)为研究对象,采用基质辅助激光解吸电离串联飞行时间质谱(MALDITOF/TOFMS)、傅里叶变换离子回旋共振质谱(FTICRMS)的碰撞活化解离(CAD)和电子转移解离(ECD)多种质谱技术对RNase B的糖肽的完整信息进行解析。通过多种质谱相互验证,确定了RNase B的糖肽组分的氨基酸序列为SRNLTK、糖链结构为(HexNAc)2(Hex)5(HexNAc)2(Hex)9及糖基化位点为第34位氨基酸残基。本方法为糖蛋白结构的全面解析提供了一种快速、有效、全面的研究思路。
关键词 基质辅助激光解吸电离串联飞行时间质谱; 傅里叶变换离子回旋共振质谱; 碰撞活化解离; 电子捕获解离; 核糖核酸酶B
1 引 言
蛋白质的糖基化是糖链在糖基转移酶作用下结合到蛋白质特定氨基酸残基上[1],是蛋白质的一种重要的翻译后修饰方式[2]。目前, 已知哺乳动物蛋白质中至少有1/2是糖蛋白,广泛分布于各种组织和细胞中[3]。糖蛋白糖链的结构多样,功能复杂,并且由于生物合成的非模板性和结构的微不均一性,使糖链具有了更多的分支结构、连接方式和空间构象,因此,修饰后的糖蛋白会产生多种生物活性,对生物体的生长、发育甚至对生物体的生存起着至关重要的作用[4]。蛋白质的糖基化类型主要为N连接和O连接的糖基化,N连接的糖基化发生在特定的氨基酸序列AsnXSer/Thr,其中X是除脯氨酸以外的任意氨基酸。O连接的糖基化发生在Ser/Thr氨基酸序列上。蛋白质糖基化与许多疾病的发展密切相关,在某些疾病状态下,聚糖的相对丰度和分支结构与正常相比发生了变化[5],这些异常的变化可为疾病的早期诊断及治疗提供帮助[6]。
常规的糖蛋白研究方法主要有基质辅助激光解吸电离飞行时间质谱(MALDITOFMS)[7]、电喷雾质谱(ESIMS)[8]、傅里叶变换离子回旋共振质谱(FTICRMS)[9]等。MALDITOFMS和ESIMS这两种方法都是对糖链和肽段分别研究,以得到糖基化位点和糖链结构信息,或者是在完整糖肽水平上对糖蛋白结构进行解析。但是在对完整糖肽进行解析时,得到的主要是糖链的结构信息,糖肽序列信息较少,导致位点鉴定困难,尤其是对于未知的样品。完整糖蛋白的全面解析对于疾病的诊断与治疗、糖蛋白药物的筛选及蛋白质组学研究具有重要意义。因此,开发完整糖肽水平的研究方法显得尤为重要。FTICRMS的碰撞诱导解离(CAD)可以得到糖链的结构信息,电子捕获解离(ECD)碎裂方式通过捕获一个自由电子,使其与质子化的多电荷肽段相互作用,从而诱发多肽主链在NCα键处发生断裂,主要产生C、Z离子,在碎片离子上保留修饰基团为翻译后修饰位点的鉴定提供了重要的信息[10,11]。MALDITOF/TOFMS样品需求量少,且提供丰富的低分子段碎片离子信息。因此,采用多重质谱的联合分析为完整糖肽的全面研究提供可能。
本研究以糖蛋白核糖核酸酶B(RNase B)的完整糖肽为研究对象,采用液相色谱分离富集,联合MALDITOF/TOFMS和FTICRMS的CAD和ECD解离模式等多种质谱技术,在解析糖链结构的同时,获得肽段序列和糖基化位点的信息,实现糖蛋白结构的全面、同步解析,为未知及复杂糖蛋白的完整糖肽的解析提供参考方法。
4 结 论
本研究采用液相色谱对样品中的糖肽进行分离富集,MALDITOF/TOFMS和FTICRMS多重质谱检测,对糖蛋白糖链结构及糖基化位点进行同步解析。MALDITOF/TOFMS通过对少量样品的完整糖肽及切除糖链的肽骨架分析,可获得肽骨架序列、糖基化位点及糖链结构的信息,分析速度快。FTICRMS的CAD碎裂模式和ECD碎裂模式相结合,不需要切除糖链即可实现肽段序列、糖基化位点及糖链结构的同时分析,如与nanoLC联用,可望进行糖肽的定量分析。多种质谱技术联合及相互验证,鉴定RNase B的糖链结构为(HexNAc)2(Hex)5(HexNAc)2(Hex)9,糖基化位点为第34位氨基酸残基,糖肽氨基酸序列为SRNLTK。本研究为复杂糖蛋白的结构解析提供了一种简单、有效的方法,对于蛋白质组学研究特别是蛋白质翻译后修饰研究具有重要的意义,同时也为糖蛋白药物的筛选及疾病的诊断与治疗提供帮助。
References
1 WANG Sheng, ZOU Xia, ZHANG Yan. Progr Chem., 2010, 22(12): 2428-2435
王 胜, 邹 霞, 张 延. 化学进展, 2010, 22(12): 2428-2435
2 SHI YuMei, ZHENG Lei, LI Juan. Progr Mod。 Biomed., 2011, 11(24): 5190-5192
石玉梅, 郑 磊, 李 娟. 现代生物医学进展, 2011, 11(24): 5190-5192
3 WANG LinJie, ZHENG DeXian, GAO Youhe. Basic Clin. Med., 2007, 27(2): 122-128
王林杰, 郑德先, 高友鹤. 基础医学与临床, 2007, 27(2): 122-128
4 XU Sha, ZHANG Ping, HUANG LinJuan, WANG ZhongFu. Chem. J. Chinese Universities, 2010, (31)10: 1992-1998
徐 莎, 张 萍, 黄琳娟, 王仲孚. 高等学校化学学报, 2010, (31)10: 1992-1998
5 CHEN Gang, BAI Quan, GENG XinDu. Chinese Journal of Chromatography, 2006, 24(5): 425-431
陈 刚, 白 泉, 耿信笃. 色谱, 2006, 24(5): 425-431
6 Alley W R, Jr, Mechref Y, Novotny M V. Rapid Commun. Mass Spectrom., 2009, 23(1): 161-170
7 Harvey D J, Royle L, Radcliffe C M, Rudd P M, Dwek R A. Anal. Biochem., 2008, 376(1): 44-60
8 Nwosu C C, Seipert R R, Strum J S, Hua S S, An H J, Zivkovic A M, German B J, Lebrilla C B. J. Proteome Res., 2011, 10(5): 2612-2624
9 Hongo Y, Nakamura T, Sato A. J. Mass Spectrom. Soc. Jpn., 2007, 55(2): 77-82
10 SUN RuiXiang, DONG MengQiu, CHI Hao, YANG Bing, XIU LiYun, WANG LeHeng, FU Yan, HE SiMin. Progr. Biochem Biophys., 2010, 37(1): 94-102
孙瑞祥, 董梦秋, 迟 浩, 杨 兵, 秀丽蕴, 王乐珩, 付 岩, 贺思敏. 生物化学与生物物理进展, 2010, 37(1): 94-102
11 LIU GuangXian, GUAN ShanHong, WANG Hui, FENG JianXiong. Food Sci., 2013, 34(17): 43-46
刘光宪, 管珊红, 王 辉, 冯健雄. 食品科学, 2013, 34(17): 43-46
12 ZENG Rong, SHAO XiaoXia, WANG KeYi, XIA QiChang. Acta Biochem. Bioph. Sinica, 1999, 31(6): 695700
曾 嵘, 邵晓霞, 王克夷, 夏其昌. 生物化学与生物物理学报, 1999, 31(6): 695-700
13 Zhao P, Viner R, Teo C F, Boons GJ, Horn D, Wells L. J. Proteome Res., 2011, 10(9): 4088-4104
14 ZHOU Wei, LIU XiaoHui, ZHOU XinWen, SHEN HuaLi, YANG PengYuan. Chinese Journal of Chromatography, 2007, 25(5): 623-627
周 玮, 刘晓慧, 周新文, 申华莉, 杨芃原. 色谱, 2007, 25(5): 623-627
15 CHEN YaoHan, YAN GuoQuan, ZHOU XinWen, YANG PengYuan. Chinese Journal of Chromatography, 2010, 28(2): 135-139
陈瑶函, 晏国全, 周新文, 杨芃原. 色谱, 2010, 28(2): 135-139
16 An H J, Peavy T R, Hedrick J L, Lebrilla C B. Anal. Chem., 2003, 75(20): 5628-5637
17 Singh C, Zampronio C G, Greese A J, Cooper H J. J. Proteome Res., 2012, 11(9): 4517-4525
18 JIANG Jing, YING WanTao, QIAN XiaoHong. Chinese J. Anal. Chem., 2014, 42(2): 159-165
江 静, 应万涛, 钱小红. 分析化学, 2014, 42(2): 159-165

