龙头鱼ISSR-PCR反应体系的建立与优化
褚梦洁 王铮 林彦均 朱兰倩 胡静雯


摘 要:以Tris平衡酚-氯仿法提取的龙头鱼基因组DNA为ISSR-PCR扩增模板,采用单因素试验法,对影响PCR扩增体系的dNTPs、模板DNA、引物浓度和Taq DNA聚合酶4种试剂的用量作为独立因素进行优化,创建了最适合龙头鱼的ISSR-PCR反应体系。结果表明:20μL的PCR反应液的组分和用量为含Mg2+的10×PCR Buffer 2.0μL,dNTPs2.5μL,Taq DNA聚合酶0.2μL,引物1.0μL,DNA模板1.4μL,双蒸水12.9μL时扩增条带最清晰。这一实验结果为龙头鱼后续开展ISSR遗传多样性分析奠定了基础。
关键词:龙头鱼;ISSR-PCR;反应条件优化
中图分类号:S917 ?文献标识码:A
龙头鱼(Harpadon nehereus Hamilton,1822)俗称狗母鱼、九肚鱼、虾潺、豆腐鱼、鼻涕鱼等,为灯笼鱼目(Myctophiformes),龙头鱼科(Harpadontidae),龙头鱼属(Harpadon)的底层中小型经济鱼类,在我国黄海南部、东海和南海均广泛分布,也是定制网和底拖网的重要渔获对象[1,2]。龙头鱼营养丰富、肉质细嫩,是受到消费者广为喜爱的海产品之一。20世纪80年代以来,随着近岸渔业资源开发力度的加大和水域环境的污染,龙头鱼种群结构及数量分布遭到了一定程度的破坏,加之龙头鱼本身存在较严重的同类自残现象,导致近年来部分海区龙头鱼群体出现低龄化和小型化问题[3],而研究龙头鱼种群遗传多样性使得我们能够发现龙头鱼群体中能够适应环境变化的个体,为保护龙头鱼群体因为自残问题而出现的低龄化和小型化问题提供解决可能性。
遗传多样性是生物多样性的重要组成部分,也是物种适应进化的基础,遗传变异越丰富,物种应对环境变化的适应能力就越强。较高水平的进化潜力也将有助于减缓因不适应进化所导致的物种灭绝过程,从而维持整个生态系统的多样性[4-5]。遺传多样性的检测手段从不同角度和层次来看,主要包括形态标记(Morphological markers)、细胞标记(Cytological markers)、生化标记(Biochemical markers)和分子标记(Molecular markers)4大类,其中分子标记能够从DNA水平揭示物种的变异程度,具有数量大、多态性高,操作简便迅速且受组织、发育阶段、季节和环境等因素的限制小等优势,成为了遗传多样性评估、品种改良与选育等领域的主要技术手段[6-7]。目前,国内外围绕龙头鱼遗传多样性开展的研究工作主要集中在线粒体DNA序列分析和核基因SSR、SRAP分子标记技术领域[8-12]。
简单重复间序列(Inter simple sequence repeat,ISSR)是在简单重复序列(Simple sequence repeats,SSR)基础上发展起来的一种新型分子标记技术,能够对整个基因组DNA进行扩增,变异丰富且多态性高,相较于SSR、RAPD等分子标记具有更好的稳定性,可以检测到更多的遗传变异[13-14]。由于ISSR-PCR反应结果受到Taq DNA聚合酶、引物、模板、dNTPs等多种因素的影响[15],因此,在利用该技术进行遗传多样性研究时,为保证结果的准确性,需要对反应体系进行优化。本研究以传统Tris平衡酚-氯仿法提取的龙头鱼基因组DNA为模板,比较分析了ISSR反应体系中dNTPs、Taq DNA聚合酶、引物和DNA模板浓度对PCR结果的影响,建立了适用于龙头鱼的ISSR最佳反应体系,为进一步开展龙头鱼分子标记研究以及其遗传多样性评价和亲缘关系分析奠定基础。
1 材料与方法
1.1 实验材料
以2019年10月采集自浙江省舟山沿海的龙头鱼为材料,剪取背部肌肉组织,采用传统的Tris平衡酚-氯仿法提取龙头鱼DNA[16],使用英国柏点(BioDrop DUO)多功能超微量分光光度计测定A260nm/A280nm值并计算DNA浓度。PCR扩增反应所用的Taq酶、引物、10×Buffer、dNTPs等试剂均购自上海生工生物工程技术服务公司。
1.2 引物筛选和退火温度确定
根据加拿大哥伦比亚大学(University of British Columbia,简称UBC)公布的100余条ISSR引物,参考在鱼类中具有较好扩增多态性的引物,设置退火温度范围在47~62℃之间的8个温度(47.0℃、48.0℃、49.8℃、52.7℃、56.2℃、58.9℃、608℃、62.0℃),梯度PCR预实验筛选出具有较高分辨率和特异性的12条ISSR引物(表1),从中再进行3~4次复筛,最终确定引物881为本实验最适引物。
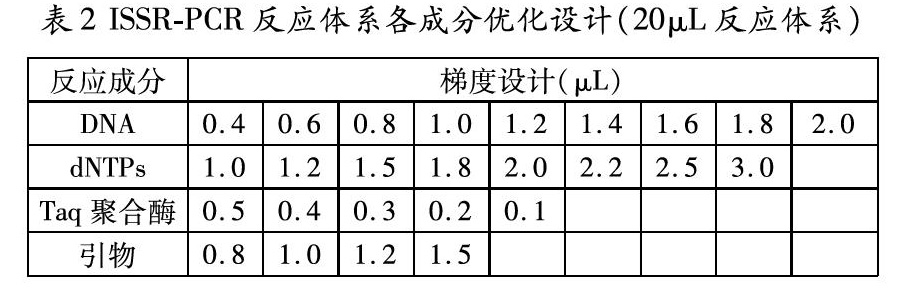
1.3 ISSR-PCR反应体系与梯度设计
PCR反应体系为20μL,包括含Mg2+的10×PCR Buffer 2μL、dNTPs 1.6μL、引物 1.6μL、Taq聚合酶0.2μL、DNA模板8μL,并用双蒸水(13.8μL)补至终体积为20μL。在此基础上,对PCR扩增中4个参数进行体积梯度设置(表2)。参考胡凤荣等[17]和曹冬梅等[18]的研究,将PCR扩增反应程序设置为:95℃预变性4min;94℃变性40s,56.2℃退火45s,72℃延伸90s,30个循环;最后72℃延伸10min,PCR产物4℃保存。每个梯度实验至少重复3次,直至筛选出扩增条带清晰、多态性和重复性好的各因素最优组合。扩增结束后,采用1%琼脂糖凝胶电泳检测并在上海天能(Tanon 2500)全自动数码凝胶图像分析仪上拍照观察。
2 结果分析
2.1 DNA模板浓度对ISSR-PCR的影响
DNA模板含量是制约扩增产物效率及特异性的一个重要因子。模板含量过低,会使分子碰撞的几率降低,没有扩增产物或扩增不稳定;模板含量过高,又会增加非特异性产物的数量[18]。图1可以看出,本研究中DNA模板浓度变化对ISSR-PCR并未产生显著的影响。当模板体积在0.8-1.8μL范围内时,扩增条带较为清晰,当体积为1.4μL和1.6μL时扩增条带最为清晰明亮,从节约实验材料考虑,故选择模板体积为1.4μL。
2.2 dNTPs濃度对ISSR-PCR的影响
dNTPs是进行PCR反应的重要原材料之一,dNTPs的浓度变化对PCR的扩增有着密不可分的关系。dNTPs浓度过高,会导致聚合酶错误掺入,产生非特异性扩增产物,过低则会造成扩增产物量少[19]。本实验设置的8个dNTPs浓度均能扩增出条带(图2),条带的清晰程度肉眼难以区别。在降低经济成本与保证清晰度的前提下,建议将dNTPs浓度调整为2.5μL,此时扩增条带最清晰且拖带较少。
2.3 Taq聚合酶浓度对ISSR-PCR的影响
Taq聚合酶浓度过高会引起非特异性扩增,浓度过低则PCR产物量减少[20]。本实验比较了0.5μL、0.4μL、0.3μL、0.2μL、0.1μL五个梯度的Taq DNA聚合酶浓度对ISSR-PCR效率的影响,结果表明五种浓度Taq DNA聚合酶均可扩增出条带,相较而言0.2μL和0.4μL时扩增条带更清晰(图3),从经济成本角度选择0.2μL Taq DNA聚合酶作为龙头鱼ISSR-PCR实验最优选择。
2.4 引物浓度对ISSR-PCR的影响
引物是PCR特异性反应的关键,浓度太低会导致扩增产量少,浓度过高又会引起错配和非特异性扩增,增加引物间形成二聚体的几率[21]。本研究中当引物体积为1.0μL时扩增条带最清晰,当体积为0.8μL时部分条带缺失且较模糊(图4)。
3 讨论
ISSP-PCR扩增受Mg2+、dNTPs、引物和DNA聚合酶等因素的影响,且各因素之间存在着相互作用。本实验中基因组DNA经电泳和分光光度计检测质量较好,在总体积20μL的反应体系中,模板DNA的浓度变化并未明显地影响实验结果,表明ISSR-PCR反应对DNA模板的用量没有严格限定。相反的,Taq聚合酶是保证实验成功的先决条件,Taq聚合酶浓度会极大地影响反应结果,浓度过高不仅不会提高扩增效率,反而容易产生非特异性扩增产物,使条带变得模糊不清。其次,过量使用Taq聚合酶也增加了经济成本,降低了实验效率。实验结果表明,dNTPs浓度与引物浓度越高,扩增条带越清晰度也越高,但由于最高浓度的扩增条带与其相邻条带间清晰度差异较小,肉眼难以区分二者差别,为节约实验成本,故选择第二高的条带。
综上所述,根据设计的变量进行多次实验,本研究最终确定了龙头鱼ISSR-PCR最优反应条件和体系,即95℃预变性4min;94℃变性40s,58.9℃退火45s,72℃延伸90s,30个循环;最后72℃延伸10min,反应产物4℃保存。PCR扩增体系采用20μL的总反应液,包含10×PCR Buffer(含Mg2+)20μL,dNTPs 2.5μL,Taq DNA聚合酶0.2μL,引物1.0μL,DNA模板1.4μL,双蒸水12.9μL。采用本研究所确定的ISSR标记的PCR体系对龙头鱼基因组DNA进行扩增,获得了清晰度高、重复性好的DNA谱带,为使用ISSR分子标记技术对龙头鱼进行种质资源鉴定和遗传多样性分析奠定了基础,也为其后续遗传学研究提供了参考资料。
参考文献:
[1]林显鹏,朱增军,李鹏飞.东海区龙头鱼摄食习性的研究.海洋渔业,2010,32(3):290-296.
[2]陈素芝.中国动物志 硬骨鱼纲:灯笼鱼目 鲸口鱼目 骨舌鱼目.北京:科学出版社,2002,83-85.
[3]陈玲,水柏年,董文霞.龙头鱼生长特征及资源的可持续利用.科技与管理,2012,(3):68-70.
[4]施立明.遗传多样性及其保存.生物科学信息,1990,2(4):159-164.
[5]季维智.遗传多样性研究的原理与方法.杭州:浙江科学技术出版社,1999.
[6]任旭琴.遗传多样性及其研究方法.淮阴工学院学报,2002,11(5):6-8.
[7]尚占环,姚爱兴.生物遗传多样性研究方法及其保护措施.农业科学研究,2002,23(1):66-69.
[8]Xu T J,Sun D Q,Li H Y,et al.Development and characterization of microsatellite markers for the lizardfish known as the Bombay duck,Harpadon nehereus(Synodontidae).Genetics & Molecular Research,2011,10(3):1701.
[9]李海燕.龙头鱼SSR和SRAP标记筛选及遗传多样性分析.舟山:浙江海洋学院,2012.
[10]Zhu Z H,Li H Y,Qin Y,et al.Genetic diversity and population structure in Harpadon nehereus based on sequence-related amplified polymorphism markers.Genetics & Molecular Research,2014,13(3):5974-5981.
[11]郭易佳,杨天燕,孟玮,等.基于线粒体Cyt b基因的龙头鱼群体遗传结构分析.水生生物学报,2019,43(5):945-952.
[12]Saha S,Ferdous Z,Jahan H,et al.Polymorphic loci analysis of 16S ribosomal RNA gene of Bombay duck(Harpadon nehereus).Bangladesh Journal of Zoology,2019,47(1):49-57.
[13]Reddy M P,Sarla N,Siddiq E A.Inter simple sequence repeat(ISSR)polymorphism and its application in plant breeding.Euphytica,2002,128(1):9-17.
[14]Souframanien J,Gopalakrishna T.A comparative analysis of genetic diversity in blackgram genotypes using RAPD and ISSR markers.Theoretical and applied genetics,2004,109(8):1687-1693.
[15]Saiki R K.The design and optimization of the PCR,in PCR Technology Erlich HA.New York:Stockton Press,1989.
[16]Sambrook J,Fritsch E F,Maniatis T.Molecular cloning:a laboratory manual.New York:Cold Spring Harbor Laboratory Press,1982.
[17]胡鳳荣,王斐,王志强,等.风信子 ISSR-PCR 体系的优化及引物筛选.分子植物育种,2013,11(1):139-141.
[18]曹冬梅,康黎芳,赵艳,等.大花萱草ISSR-PCR最佳反应体系的建立.山西农业科学,2011,39(12):1239-1242.
[19]卢盛栋.分子生物学实验.中国协和医科大学出版社,1999:458-463.
[20]蒋兴川,和淑琪,杨明,等.西花蓟马ISSR-PCR反应体系的建立与优化.生物安全学报,2011,20(3):198-206.
[21]余艳,陈海山,葛学军.简单重复序列区间(ISSR)引物反应条件优化与筛选.热带亚热带植物学报,2003,11(1):15-19.
基金项目:浙江省大学生科技创新活动计划暨新苗人才计划项目(2019R411013);浙江海洋大学本科生发展性资助项目;浙江海洋大学“水产”省一流学科大学生创新性科研项目(11034060216)
作者简介:褚梦洁(1999—),女,汉族,浙江萧山人,本科,研究方向:海洋资源与环境。

